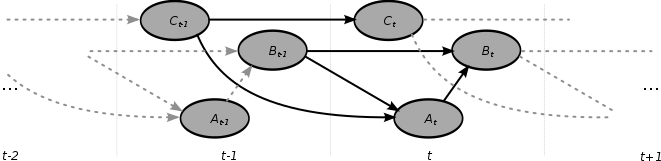
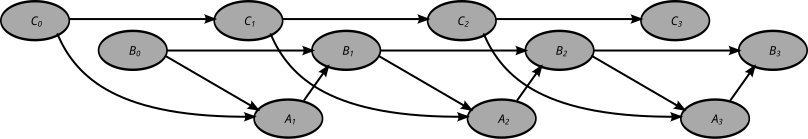
Réseau bayésien dynamique - Dynamic Bayesian network
Un réseau bayésien dynamique (DBN) est un réseau bayésien (BN) qui relie les variables les unes aux autres sur des pas de temps adjacents. Ceci est souvent appelé un BN à deux tranches temporelles (2TBN) car il indique qu'à tout moment T, la valeur d'une variable peut être calculée à partir des régresseurs internes et de la valeur immédiatement antérieure (temps T-1). Les DBN ont été développés par Paul Dagum au début des années 90 à la section d'informatique médicale de l'université de Stanford . Dagum a développé des DBN pour unifier et étendre les modèles d'espace d'état linéaires traditionnels tels que les filtres de Kalman , les modèles de prévision linéaires et normaux tels que ARMA et les modèles de dépendance simples tels quemodèles de Markov cachés en une représentation probabiliste générale et un mécanisme d'inférence pour des domaines arbitraires non linéaires et non normaux dépendant du temps.
Aujourd'hui, les DBN sont courants en robotique et ont montré un potentiel pour un large éventail d' applications d' exploration de données . Par exemple, ils ont été utilisés dans la reconnaissance vocale , la criminalistique numérique , le séquençage des protéines et la bioinformatique . DBN est une généralisation des modèles de Markov cachés et des filtres de Kalman .
Les DBN sont conceptuellement liés aux réseaux booléens probabilistes et peuvent, de la même manière, être utilisés pour modéliser des systèmes dynamiques en régime permanent.
Voir également
Les références
Lectures complémentaires
- Murphy, Kevin (2002). Réseaux bayésiens dynamiques : représentation, inférence et apprentissage . UC Berkeley, division informatique.
- Ghahramani, Zoubin (1997). Apprentissage des réseaux bayésiens dynamiques . Notes de cours en informatique . 1387 . p. 168-197. CiteSeerX 10.1.1.56.7874 . doi : 10.1007/BFb0053999 . ISBN 978-3-540-64341-8.
- Friedman, N.; Murphy, K.; Russell, S. (1998). Apprentissage de la structure des réseaux probabilistes dynamiques . AUI'98. Morgan Kaufmann. p. 139-147. CiteSeerX 10.1.1.75.2969 .
Logiciel
- bnt sur GitHub : la Bayes Net Toolbox for Matlab, par Kevin Murphy, (publié sous licence GPL )
- Graphical Models Toolkit (GMTK) : une boîte à outils open source et accessible au public pour le prototypage rapide de modèles statistiques à l'aide de modèles graphiques dynamiques (DGM) et de réseaux bayésiens dynamiques (DBN). GMTK peut être utilisé pour des applications et des recherches dans le traitement de la parole et du langage, la bioinformatique, la reconnaissance d'activité et toute application de séries chronologiques.
- DBmcmc : Inferring Dynamic Bayesian Networks with MCMC, pour Matlab (logiciel gratuit)
- Boîte à outils GlobalMIT Matlab chez Google Code : Modélisation du réseau de régulation des gènes via l'optimisation globale du réseau bayésien dynamique (publié sous licence GPL )
- libDAI : bibliothèque C++ qui fournit des implémentations de diverses méthodes d'inférence (approximatives) pour des modèles graphiques discrets ; prend en charge les graphiques à facteurs arbitraires avec des variables discrètes, y compris les champs aléatoires de Markov discrets et les réseaux bayésiens (publiés sous la licence FreeBSD )
- aGrUM : bibliothèque C++ (avec liaisons Python) pour différents types de PGM, y compris les réseaux bayésiens et les réseaux bayésiens dynamiques (publié sous la GPLv3)
- FALCON : Boîte à outils Matlab pour la contextualisation des modèles DBN de réseaux de régulation avec des données quantitatives biologiques, incluant divers schémas de régularisation pour modéliser les connaissances biologiques antérieures (publié sous la GPLv3)