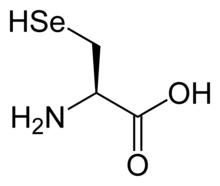
Sélénocystéine - Selenocysteine
|
|
| Noms | |
|---|---|
|
Nom IUPAC
Acide 2-amino-3-sélanylpropanoïque
|
|
| Autres noms
L- Sélénocystéine; 3-Sélanyl- L -alanine ; Sélénium cystéine
|
|
| Identifiants | |
|
Modèle 3D ( JSmol )
|
|
| ChEBI | |
| ChEMBL | |
| ChemSpider | |
| Banque de médicaments | |
| Carte d'information de l'ECHA |
100.236.386 |
| KEGG | |
|
CID PubChem
|
|
| UNII | |
|
Tableau de bord CompTox ( EPA )
|
|
|
|
|
|
| Propriétés | |
| C 3 H 7 N O 2 Se | |
| Masse molaire | 168,065 g·mol -1 |
|
Sauf indication contraire, les données sont données pour les matériaux dans leur état standard (à 25 °C [77 °F], 100 kPa). |
|
|
|
|
| Références de l'infobox | |
La sélénocystéine (symbole Sec ou U , dans les publications plus anciennes également sous le nom de Se-Cys ) est le 21e acide aminé protéinogène . Les sélénoprotéines contiennent des résidus de sélénocystéine. La sélénocystéine est un analogue de la cystéine plus courante avec du sélénium à la place du soufre .
La sélénocystéine est présente dans plusieurs enzymes (par exemple les glutathion peroxydases , les tétraiodothyronine 5′ déiodinases , les thiorédoxines réductases , les formiates déshydrogénases , les glycine réductases , la sélénophosphate synthétase 2 , la méthionine- R- sulfoxyde réductase B1 ), et certaines hydrogénases SEPX1 . Il se produit dans les trois domaines de la vie .
La sélénocystéine a été découverte par la biochimiste Thressa Stadtman aux National Institutes of Health .
Chimie
La sélénocystéine est l'analogue Se de la cystéine. Il est rarement rencontré (et non disponible dans le commerce) car il est très sensible à l'oxydation par l'air. Le dérivé oxydé sélénocystine, qui a une liaison Se-Se, est plus courant. La sélénocystéine et la sélénocystine sont toutes deux des solides blancs. Le groupe Se-H est plus acide ( p K a = 5,43) que le groupe thiol, il est donc déprotoné au pH physiologique .
Structure
La sélénocystéine a une structure similaire à celle de la cystéine , mais avec un atome de sélénium prenant la place du soufre habituel. Il a un groupe sélénol . Comme d'autres acides aminés protéinogènes naturels, la cystéine et la sélénocystéine ont une chiralité L dans l'ancienne notation D / L basée sur une homologie avec le D- et L - glycéraldéhyde . Dans le nouveau système R / S de désignation de la chiralité, basé sur les numéros atomiques des atomes proches du carbone asymétrique, ils ont une chiralité R , en raison de la présence de soufre ou de sélénium comme deuxième voisin du carbone asymétrique. Les acides aminés chiraux restants, n'ayant que des atomes plus légers dans cette position, ont une chiralité S. )
Les protéines qui contiennent un résidu sélénocystéine sont appelées sélénoprotéines . La plupart des sélénoprotéines contiennent un seul résidu de sélénocystéine. Les sélénoprotéines qui présentent une activité catalytique sont appelées sélénoenzymes.
La biologie
La sélénocystéine a un potentiel de réduction inférieur à celui de la cystéine. Ces propriétés le rendent très approprié dans les protéines impliquées dans l' activité antioxydante .
Bien qu'on le retrouve dans les trois domaines de la vie, il n'est pas universel dans tous les organismes. Contrairement aux autres acides aminés présents dans les protéines biologiques , la sélénocystéine n'est pas codée directement dans le code génétique . Au lieu de cela, il est codé d'une manière spéciale par un codon UGA , qui est normalement le codon stop "opale" . Un tel mécanisme est appelé recodage traductionnel et son efficacité dépend de la sélénoprotéine synthétisée et des facteurs d'initiation de la traduction . Lorsque les cellules sont cultivées en l'absence de sélénium, la traduction des sélénoprotéines se termine au niveau du codon UGA, résultant en une enzyme tronquée et non fonctionnelle. Le codon UGA est fait pour coder la sélénocystéine par la présence d'une séquence d'insertion de sélénocystéine (SECIS) dans l' ARNm . L'élément SECIS est défini par des séquences nucléotidiques caractéristiques et des motifs d'appariement de bases de structure secondaire. Dans les bactéries , l'élément SECIS est généralement situé immédiatement après le codon UGA dans le cadre de lecture de la sélénoprotéine. Chez les archées et les eucaryotes , l'élément SECIS se trouve dans la région 3' non traduite (3' UTR) de l'ARNm et peut diriger plusieurs codons UGA pour coder les résidus sélénocystéine.
Contrairement aux autres acides aminés, aucun pool libre de sélénocystéine n'existe dans la cellule. Sa réactivité élevée causerait des dommages aux cellules. Au lieu de cela, les cellules stockent le sélénium sous la forme oxydée moins réactive, la sélénocystine, ou sous la forme méthylée, la sélénométhionine. La synthèse de la sélénocystéine se produit sur un ARNt spécialisé , qui fonctionne également pour l'incorporer dans des polypeptides naissants.
La structure primaire et secondaire de l'ARNt spécifique de la sélénocystéine, l'ARNt Sec , diffère de celles des ARNt standard à plusieurs égards, notamment en ce qu'elle possède une tige acceptrice de 8 paires de bases (bactéries) ou de 10 paires de bases (eucaryotes), un bras de région variable long et substitutions à plusieurs positions de base bien conservées. Les ARNt de sélénocystéine sont initialement chargés en sérine par la séryl-ARNt ligase , mais le Ser-ARNt Sec résultant n'est pas utilisé pour la traduction car il n'est pas reconnu par le facteur d'élongation de la traduction normale ( EF-Tu chez les bactéries, eEF1A chez les eucaryotes).
Au lieu de cela, le résidu séryle lié à l'ARNt est converti en un résidu sélénocystéine par l' enzyme sélénocystéine synthase contenant du phosphate de pyridoxal . Chez les eucaryotes et les archées, deux enzymes sont nécessaires pour convertir le résidu séryle lié à l'ARNt en résidu sélénocystéinyle d'ARNt : PSTK ( O -phosphoseryl-tRNA[Ser]Sec kinase) et sélénocystéine synthase. Enfin, le Sec-ARNt Sec résultant est spécifiquement lié à un facteur d'élongation traductionnel alternatif (SelB ou mSelB (ou eEFSec)), qui le délivre de manière ciblée aux ribosomes traduisant les ARNm des sélénoprotéines. La spécificité de ce mécanisme de délivrance est provoquée par la présence d'un domaine protéique supplémentaire (chez les bactéries, SelB) ou d'une sous-unité supplémentaire ( SBP2 pour eucaryote mSelB/eEFSec) qui se lient aux structures secondaires d'ARN correspondantes formées par les éléments SECIS dans la sélénoprotéine. ARNm.
La sélénocystéine est décomposée par l'enzyme sélénocystéine lyase en L - alanine et séléniure.
En 2021, 136 protéines humaines (dans 37 familles) sont connues pour contenir de la sélénocystéine (sélénoprotéines).
Les dérivés de la sélénocystéine γ-glutamyl- Se- méthylsélénocystéine et Se- méthylsélénocystéine sont naturellement présents dans les plantes des genres Allium et Brassica .
Applications
Applications biotechnologiques de sélénocystéine comprennent l' utilisation de 73 Se-marqué Sec (demi-vie de 73 Se = 7,2 heures) dans la tomographie par émission de positrons (PET) des études et 75 Se-marqué Sec (demi-vie de 75 Se = 118,5 jours) en particulier radiomarquage , facilitation de la détermination de phase par diffraction anormale à plusieurs longueurs d'onde dans la cristallographie aux rayons X des protéines en introduisant Sec seul, ou Sec avec la sélénométhionine (SeMet), et incorporation de l'isotope 77 Se stable , qui a un spin nucléaire de1/2et peut être utilisé pour la RMN haute résolution , entre autres.
Voir également
- Pyrrolysine , un autre acide aminé qui ne fait pas partie de l'ensemble de base de 20.
- La sélénométhionine , un autre acide aminé contenant du sélénium, qui est substitué au hasard à la méthionine.
Les références
Lectures complémentaires
- Zinoni F, Birkmann A, Stadtman TC, Böck A (juillet 1986). "Séquence nucléotidique et expression du polypeptide contenant de la sélénocystéine de la formiate déshydrogénase (liée au formiate-hydrogène-lyase) d'Escherichia coli" . Actes de l'Académie nationale des sciences des États-Unis d'Amérique . 83 (13) : 4650-4. Bibcode : 1986PNAS ... 83.4650Z . doi : 10.1073/pnas.83.13.4650 . PMC 323799 . PMID 2941757 .
- Zinoni F, Birkmann A, Leinfelder W, Böck A (mai 1987). « Insertion cotraductionnelle de la sélénocystéine dans la formiate déshydrogénase d'Escherichia coli dirigée par un codon UGA » . Actes de l'Académie nationale des sciences des États-Unis d'Amérique . 84 (10) : 3156–60. Bibcode : 1987PNAS ... 84.3156Z . doi : 10.1073/pnas.84.10.3156 . PMC 304827 . PMID 3033637 .
- Cone JE, Del Río RM, Davis JN, Stadtman TC (août 1976). « Caractérisation chimique du composant sélénoprotéique de la glycine réductase clostridienne : identification de la sélénocystéine en tant que partie organosélénium » . Actes de l'Académie nationale des sciences des États-Unis d'Amérique . 73 (8) : 2659-63. Bibcode : 1976PNAS ... 73.2659C . doi : 10.1073/pnas.73.8.2659 . PMC 430707 . PMID 1066676 .
- Fenyö D, Beavis RC (février 2016). "Sélénocystéine : pourquoi es-tu ?". Journal de recherche sur le protéome . 15 (2) : 677-8. doi : 10.1021/acs.jproteome.5b01028 . PMID 26680273 .
Liens externes
| Scholia a un profil de sujet pour la sélénocystéine . |
