Atromentine - Atromentin
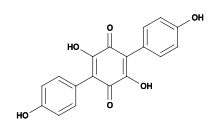 Formule développée de l'atromentine
|
|
| Des noms | |
|---|---|
|
Nom IUPAC préféré
1 4 , 2 3 , 2 6 , 3 4 -Tétrahydroxy [1 1 , 2 1 : 2 4 , 3 1 -triphényl] -2 2 , 2 5 -dione |
|
| Autres noms
2,5-dihydroxy-3,6-bis (4-hydroxyphényl) -1,4-benzoquinone
|
|
| Identifiants | |
|
Modèle 3D ( JSmol )
|
|
| ChEBI | |
| ChemSpider | |
|
PubChem CID
|
|
| UNII | |
|
Tableau de bord CompTox ( EPA )
|
|
|
|
|
|
| Propriétés | |
| C 18 H 12 O 6 | |
| Masse molaire | 324,288 g · mol −1 |
|
Sauf indication contraire, les données sont données pour les matériaux dans leur état standard (à 25 ° C [77 ° F], 100 kPa). |
|
|
|
|
| Références Infobox | |
L'atromentine est un composé chimique naturel présent dans les champignons Agaricomycetes dans les ordres Agaricales et Thelephorales . Il peut également être préparé par synthèse en laboratoire . Chimiquement, c'est un polyphénol et une benzoquinone .
Occurrences
L'atromentine a été trouvée dans des cultures de Clitocybe subilludens et dans des extraits d' Hydnellum peckii . Les premières enzymes de sa biosynthèse ont été caractérisées chez Tapinella panuoides . L'un de ceux-ci s'appelle l' atromentine synthétase .
Activités biologiques
Un certain nombre d' actifs biologiques in vitro d'atromentine ont été étudiés. Atromentin possède in vitro une activité antibactérienne, inhibant l'enzyme protéine porteuse énoyl-acyl reductase (essentiel pour la biosynthèse des acides gras ) dans les bactéries Streptococcus pneumoniae . Il a été démontré que l'atromentine est un stimulant des muscles lisses . Il induit également l' apoptose dans les cellules isolées de leucémie humaine U937 . C'est aussi un anticoagulant .
Base génétique et enzymatique de l'atromentine
L'atromentine est catalysée à partir de deux unités d'acide 4-hydroxyphénylpyruvique (4-HPP) via une enzyme de type peptide synthétase non tribosomale (atromentine synthétase), contenant l'architecture de domaine adénylation-thiolation-thioestérase (AT-TE). La 4-HPP est produite à partir d'une désamination via une aminotransférase. La base génétique de ces deux gènes est groupée (c'est-à-dire adjacente l'un à l'autre). Ces enzymes ont d'abord été caractérisées chez Tapinella panuoides en surexprimant les gènes respectifs (AtrA et AtrD) chez E. coli et en incubant l'holo-enzyme avec la 4-HPP pour observer la formation d'atromentine. Ceci a été suivi par la caractérisation de l'enzyme GreA chez Suillus grevillei , six (InvA1-6, dont InvA1, 2 et 5 étaient fonctionnels) chez Paxillus involutus et NPS3 de Serpula lacrymans . De plus, il existe un autre gène adjacent et conservé codant pour une alcool déshydrogénase / oxydoréductase dont la fonction n'est pas claire. Dans la plupart des cas, les gènes biosynthétiques groupés se trouvent orthologues chez les basidiomycètes. Un motif promoteur commun a été trouvé partagé entre l'atromentine synthétase et l'aminotransférase de 23 basidiomycètes producteurs d'atromentine différents qui étaient dans presque tous les cas absents de l'alcool déshydrogénase, indiquant une co-régulation des deux gènes essentiels qui assurent la production d'atromentine par un facteur de transcription commun . Des motifs promoteurs supplémentaires ont été identifiés avant les gènes de l'atromentine pour les ectomycorrihzae qui étaient absents des pourritures brunes, indiquant une régulation génétique différente de l'atromentine. Les gènes de l'atromentine synthétase et de l'aminotransférase de S. lacrymans ont été régulés à la hausse pendant la co-incubation avec des bactéries.
Code nonribosomique des acides aminés pour la biosynthèse
L'enzyme de type peptide synthétase non tribosomale (atromentine synthétase) qui condense symétriquement deux monomères de 4-HPP a un domaine d'adénylation qui accepte les substrats avant la catalyse. Le domaine accepteur contient un code de 10 acides aminés connu sous le nom de code non tribosomal (code NRPS). Ici, l'exemple de l'atromentine synthétase de Suills grevillei , GreA, est utilisé. Le code se trouve aux positions d'acides aminés 235 (V), 236 (A), 239 (E), 278 (F), 299 (S), 301 (G), 322 (G), 320 (A), 331 ( C), 517 (K). Le code s'aligne avec l'atromentine synthétases de S. lacrymans (NPS3), Tapinella panuoides (AtrA) et Paxillus involutus (InvAs). De même, le code NRPS pour la production d'atromentine prend en charge le code universel pour d'autres composés dérivés de l'acide alpha-céto aromatique, tels que ceux de la L -phénylalanine comme la ralfuranone B via l'acide phénylpyruvique et du L -tryptophane comme la didéméthylastérriquinone D via l' indole-3- acide pyruvique (notez que l'atromentine est dérivée de l'acide alpha-céto aromatique L-tyrosine via l'acide 4-hydroxyphénylpyruvique).
Pour les InvAs de Paxillus involutus , un motif d'acide aminé commun a également été trouvé dans le domaine de la thioestérase (dernier domaine) qui a soutenu les données biochimiques de l'enzyme étant fonctionnelle pour compléter la formation d'atromentine ou non.
Biosynthèse de l'atromentine
L'acide aminé aromatique L -tyrosine est le précurseur de l'acide 4-hydroxyphénylpyruvique, et 2 unités de 4-HPP sont condensées pour former de l'atromentine. La première étape est la désamination via une aminotransférase. La deuxième étape est catalysée par une enzyme de type peptide synthétase non tribosomale (de type NRPS, car elle ne possède pas de domaine de condensation canonique, appelé atromentine / quinone synthétase). Le domaine d'adénylation de cette enzyme de type NRPS accepte la 4-HPP comme déterminé par le test d'échange ATP-PPi. L'enzyme, lorsqu'elle est produite dans E. coli , doit être amorcée à sa forme holo via une phosphopantetheinyl transférase (Ppant), bien que E. coli puisse in vivo amorcer l'apo-enzyme (par exemple via EntD). Les Ppants ont été utilisés avec succès à partir d'ADNc dérivé de A. nidulans (par exemple NpgA), Streptomyces verticillus (Svp) et Paxillus involutus (PptA). Quelques études, notamment à partir de la bactérie Burkholderia thailandensis de Biggins et al., Ont montré que le gène de l'aminotransférase peut être absent et que cette activité peut être fournie via son métabolisme primaire.
Pigments de congénères
L'atromentine est le précurseur de divers autres pigments. Famille de type acide pulvinique comprennent l' acide variegatic , l' acide xerocomic , l' acide homoxerocomic , l' acide isoxerocomic , l' acide atromentic , variegatorubin , xerocomorubin , et d' autres dérivés modifiés. Les principaux pigments de type acide pulvinique ont été trouvés sécrétés lors de la co-incubation avec des bactéries ou de l'introduction à haute teneur en azote organique (par rapport à la croissance sur un milieu non inducteur contenant de l'azote inorganique). Les diarylcyclopenténones comprennent l'involutine, l'involuton, la gyrocycanine, la gyroporine (variante oxydée de la gyrocyanine), l'anhydroinvolutine et la chamonixine. Bien que structurellement similaires, les grevillines (AD) sont dérivées de la 4-HPP, le précurseur de l'atromentine. Les grévillines sont un marqueur chimiotaxonomique du genre Suillus. Les modifications de l'atromentine comprennent la leucoatromentine, la leucomentine-3, la leucomentine-4 et la cylcoleucomélone. De plus, l'acide théléphorique est un dérivé du clade théléphoroïde. Les différentes enzymes impliquées dans la formation de ces pigments en dehors de la base génétique et enzymatique de la production de son précurseur atromentine sont inconnues.
Biosynthèse redondante
Dans Paxillus involutus , six enzymes de type peptide synthétase non tribosomales ont été identifiées dans le génome annoté disponible via le portail JGI MycoCosm. Ces gènes, appelés InvA1,2,3,4,5 et 6, ont été surexprimés dans E. coli et les gènes ont été caractérisés en co-incubant l'apo-enzyme avec 4-HPP pour déterminer la formation d'atromentine comme indiqué par sa caractéristique Spectre UV-Vis et masse monoisotopique. Trois des six enzymes se sont révélées fonctionnelles. Cela a montré une redondance sans précédent pour la production d'atromentine dans un basidiomycète.
