Informatique Bioimage - Bioimage informatics
L'informatique de la bioimage est un sous-domaine de la bioinformatique et de la biologie computationnelle . Il se concentre sur l'utilisation de techniques informatiques pour analyser les bio-images, en particulier les images cellulaires et moléculaires, à grande échelle et à haut débit. L'objectif est d'obtenir des connaissances utiles à partir d'images complexes et hétérogènes et de métadonnées associées .
Les microscopes automatisés sont capables de collecter un grand nombre d'images avec une intervention minimale. Cela a conduit à une explosion des données, qui nécessite absolument un traitement automatique. De plus, et étonnamment, pour plusieurs de ces tâches, il est prouvé que les systèmes automatisés peuvent fonctionner mieux que les humains. De plus, les systèmes automatisés sont impartiaux, contrairement à l'analyse humaine dont l'évaluation peut (même inconsciemment) être influencée par le résultat souhaité.
L'accent a été mis de plus en plus sur le développement de nouvelles techniques de traitement d'images , de vision par ordinateur , d' exploration de données , de bases de données et de visualisation pour extraire, comparer, rechercher et gérer les connaissances biologiques dans ces problèmes à forte intensité de données.
Modalités de données
Plusieurs systèmes et plates-formes de collecte de données sont utilisés, qui nécessitent différentes méthodes pour être traitées de manière optimale.
Microscopie fluorescente
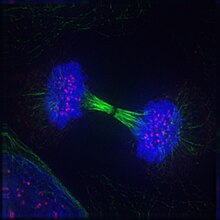
La microscopie fluorescente permet la visualisation directe des molécules au niveau subcellulaire, à la fois dans les cellules vivantes et fixes . Les molécules d'intérêt sont marquées avec une protéine fluorescente verte (GFP), une autre protéine fluorescente ou un anticorps marqué par fluorescence . Plusieurs types de microscopes sont régulièrement utilisés: champ large, confocal ou à deux photons . La plupart des systèmes de microscopie prendront également en charge la collecte de séries chronologiques (films).
En général, les filtres sont utilisés de sorte que chaque colorant est imagé séparément (par exemple, un filtre bleu est utilisé pour l'image Hoechst , puis rapidement commuté vers un filtre vert pour l'image GFP). Pour la consommation, les images sont souvent affichées en fausses couleurs en montrant chaque canal dans une couleur différente, mais celles-ci peuvent même ne pas être liées aux longueurs d'onde d'origine utilisées. Dans certains cas, l'image d'origine peut même avoir été acquise dans des longueurs d'onde non visibles (l'infrarouge est courant).
Les choix au stade de l'acquisition d'image influeront sur l'analyse et nécessitent souvent un traitement spécial. Les piles confocales nécessiteront un traitement 3D et les pseudo-piles à champ large bénéficieront souvent d' une déconvolution numérique pour éliminer la lumière floue .
L'avènement des microscopes automatisés capables d'acquérir automatiquement de nombreuses images est l'une des raisons pour lesquelles l'analyse ne peut pas être effectuée à l'œil nu (sinon, l'annotation deviendrait rapidement le goulot d'étranglement de la recherche). L'utilisation de microscopes automatisés signifie que certaines images peuvent être floues (les systèmes de recherche automatique de mise au point peuvent parfois être incorrects), contenir un petit nombre de cellules ou être remplies de débris. Par conséquent, les images générées seront plus difficiles à analyser que les images acquises par un opérateur car ils auraient choisi d'autres emplacements pour l'image et la mise au point correctement. En revanche, l'opérateur peut introduire un biais inconscient dans sa sélection en ne choisissant que les cellules dont le phénotype est le plus proche de celui attendu avant l'expérience.
Histologie
L'histologie est une application de microscopie dans laquelle des tranches de tissu sont colorées et observées au microscope (généralement au microscope optique, mais la microscopie électronique est également utilisée).
Lors de l'utilisation d'un microscope optique, contrairement au cas de l'imagerie fluorescente, les images sont généralement acquises à l'aide de systèmes de caméras couleur standard. Cela reflète en partie l'histoire du terrain, où les humains interprétaient souvent les images, mais aussi le fait que l'échantillon peut être éclairé avec de la lumière blanche et toute la lumière collectée plutôt que d'avoir à exciter les fluorophores. Lorsque plus d'un colorant est utilisé, une étape de prétraitement nécessaire consiste à démélanger les canaux et à récupérer une estimation des intensités spécifiques du colorant pur.
Il a été montré que l'emplacement subcellulaire des protéines colorées peut être identifié à partir d'images histologiques.
Si l'objectif est un diagnostic médical, alors les applications d'histologie tomberont souvent dans le domaine de la pathologie numérique ou de l'analyse automatisée d'images tissulaires , qui sont des domaines sœurs de l'informatique de la bioimage. Les mêmes techniques de calcul sont souvent applicables, mais les objectifs sont davantage axés sur la médecine que sur la recherche.
Problèmes importants
Analyse de l'emplacement subcellulaire

L'analyse de localisation subcellulaire a été l'un des problèmes initiaux dans ce domaine. Dans son mode supervisé, le problème est d'apprendre un classificateur capable de reconnaître les images des principaux organites cellulaires à partir d'images.
Les méthodes utilisées sont basées sur l'apprentissage automatique , construisant un classificateur discriminant basé sur des caractéristiques numériques calculées à partir de l'image. Les caractéristiques sont soit des caractéristiques génériques issues de la vision par ordinateur , telles que des caractéristiques de texture Haralick, soit des caractéristiques spécialement conçues pour capturer des facteurs biologiques (par exemple, la colocalisation avec un marqueur nucléaire étant un exemple typique).
Pour le problème de base de l'identification des organites, des valeurs de précision très élevées peuvent être obtenues, y compris mieux que? résultats. Ces méthodes sont utiles dans la recherche fondamentale en biologie cellulaire, mais ont également été appliquées à la découverte de protéines dont l'emplacement change dans les cellules cancéreuses.
Cependant, la classification en organites est une forme limitée du problème car de nombreuses protéines se localiseront simultanément à plusieurs endroits (motifs mixtes) et de nombreux motifs peuvent être distingués même s'ils ne sont pas des composants liés à la membrane différents. Il existe plusieurs problèmes non résolus dans ce domaine et des recherches sont en cours.
Projection à haut contenu
Les écrans à haut débit utilisant la technologie d'imagerie automatisée (parfois appelée criblage à haut contenu ) sont devenus une méthode standard à la fois pour la découverte de médicaments et la recherche biologique fondamentale. En utilisant des plaques multi-puits, la robotique et la microscopie automatisée, le même test peut être appliqué à une grande bibliothèque de réactifs possibles (généralement de petites molécules ou de l' ARNi ) très rapidement, obtenant des milliers d'images en peu de temps. En raison du volume élevé de données générées, l'analyse automatique des images est une nécessité.
Lorsque des contrôles positifs et négatifs sont disponibles, le problème peut être abordé comme un problème de classification et les mêmes techniques de calcul de caractéristiques et de classification qui sont utilisées pour l'analyse de localisation subcellulaire peuvent être appliquées.
Segmentation
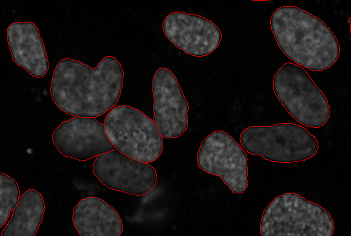
La segmentation des cellules est un sous-problème important dans de nombreux domaines ci-dessous (et parfois utile en soi si le but est uniquement d'obtenir un nombre de cellules dans un test de viabilité ). Le but est d'identifier les limites des cellules dans une image multicellulaire. Cela permet de traiter chaque cellule individuellement pour mesurer les paramètres. Dans les données 3D, la segmentation doit être effectuée dans l'espace 3D.
Comme l'imagerie d'un marqueur nucléaire est commune à de nombreuses images, un protocole largement utilisé consiste à segmenter les noyaux. Cela peut être utile en soi si des mesures nucléaires sont nécessaires ou cela peut servir à amorcer un bassin versant qui étend la segmentation à toute l'image.
Toutes les principales méthodes de segmentation ont été rapportées sur des images de cellules, du simple seuillage aux méthodes de réglage de niveau. Parce qu'il existe plusieurs modalités d'image et différents types de cellules, dont chacun implique des compromis différents, il n'y a pas de solution unique acceptée pour ce problème.
La segmentation d'image cellulaire en tant que procédure importante est souvent utilisée pour étudier l'expression génique et la relation de colocalisation, etc. de cellules individuelles. Dans de tels cas d'analyse de cellule unique, il est souvent nécessaire de déterminer de manière unique les identités des cellules tout en segmentant les cellules. Une telle tâche de reconnaissance est souvent non triviale en termes de calcul. Pour les organismes modèles tels que C. elegans qui ont des lignées cellulaires bien définies, il est possible de reconnaître explicitement les identités cellulaires via l'analyse d'image, en combinant à la fois des méthodes de segmentation d'image et de reconnaissance de formes. La segmentation et la reconnaissance simultanées des cellules ont également été proposées comme solution plus précise pour ce problème lorsqu'un «atlas» ou d'autres informations antérieures de cellules sont disponibles. Étant donné que l'expression génique à une résolution de cellule unique peut être obtenue en utilisant ces types d'approches basées sur l'imagerie, il est possible de combiner ces méthodes avec d'autres méthodes de quantification d'expression de gène à cellule unique telles que RNAseq.
Suivi
Le suivi est un autre problème de traitement d'image traditionnel qui apparaît dans l'informatique de la bioimage. Le problème est de relier les objets qui apparaissent dans les images suivantes d'un film. Comme pour la segmentation, le problème peut être posé à la fois sous des formes bidimensionnelles et tridimensionnelles.
Dans le cas de l'imagerie fluorescente, le suivi doit souvent être effectué sur des images à très faible contraste. Comme l'obtention d'un contraste élevé se fait en faisant briller plus de lumière qui endommage l'échantillon et détruit le colorant , l'éclairage est maintenu à un minimum. Il est souvent utile de penser à un budget photonique: le nombre de photons pouvant être utilisés pour l'imagerie avant les dommages à l'échantillon est si grand que les données ne peuvent plus être fiables. Par conséquent, si des images à contraste élevé doivent être obtenues, seules quelques images peuvent être utilisées; tandis que pour les longs films, chaque image aura un contraste très faible.
Inscription
Lorsque des échantillons de données d'image de natures différentes, tels que ceux correspondant à différentes méthodes d'étiquetage, à différents individus, à des échantillons à différents moments, etc. sont considérés, les images doivent souvent être enregistrées pour une meilleure comparaison. Un exemple est que lorsque les données de cours du temps sont collectées, les images dans les images suivantes doivent souvent être enregistrées afin que des changements mineurs dans la position de la caméra puissent être corrigés. Un autre exemple est que lorsque de nombreuses images d'un animal modèle (par exemple C. elegans ou Drosophila cerveau ou un cerveau de souris ) sont collectées, il est souvent nécessaire d'enregistrer ces images pour comparer leurs modèles (par exemple, ceux qui correspondent à la même population de neurones, ceux-ci partagent ou diffèrent dans l'expression génique, etc.).
Les progiciels d'enregistrement d'images médicales ont été les premières tentatives d'utilisation pour les applications d'enregistrement d'images microscopiques. Cependant, en raison de la taille souvent beaucoup plus grande du fichier image et du nombre beaucoup plus important de spécimens dans les expériences, il est souvent nécessaire de développer un nouveau logiciel d'enregistrement d'images 3D. Le BrainAligner [12] est un logiciel qui a été utilisé pour automatiser le processus d'enregistrement 3D déformable et non linéaire en utilisant une stratégie d'appariement de points de repère fiable. Il a été principalement utilisé pour générer plus de 50000 images cérébrales de mouches des fruits standardisées en 3D à Janelia Farm of HHMI, avec d'autres applications, notamment des libellules et des souris.
Lieux importants
Un consortium de scientifiques d'universités et d'instituts de recherche organise des réunions annuelles sur l'informatique de la bioimage depuis 2005. La conférence ISMB a une piste Bioimaging & Data Visualization depuis 2010. La revue Bioinformatics a également introduit une piste Bioimage Informatics en 2012. La revue OpenAccess BMC Bioinformatics a une section consacrée à l'analyse de bioimage, à la visualisation et aux applications associées. D'autres revues de biologie computationnelle et de bioinformatique publient également régulièrement des travaux d'informatique sur la bioimage. Une action de l'Union européenne sur les coûts appelée NEUBIAS (réseau des analystes européens de la bioimage) organise des conférences annuelles ainsi que des écoles de formation d'analystes en bioimage et des taggathons depuis 2017.
Logiciel
Il existe plusieurs packages qui rendent les méthodes informatiques de bioimage disponibles via une interface utilisateur graphique telle que ImageJ , FIJI , CellProfiler ou Icy . Des plates-formes de visualisation et d'analyse telles que Vaa3D sont apparues ces dernières années et ont été utilisées dans les deux projets à grande échelle, en particulier pour les neurosciences et les applications de bureau.
D'autres chercheurs développent leurs propres méthodes, généralement basées sur un langage de programmation avec une bonne prise en charge de la vision par ordinateur comme Python , C ++ ou MATLAB . La bibliothèque Mahotas pour Python est un exemple populaire. Cependant, des exemples de méthodes développées par des chercheurs dans des langages de programmation avec moins de support de vision par ordinateur que R existent (par exemple trackdem).
Voir également
- Empilement de mise au point Technique de combinaison de plusieurs images avec des distances de mise au point différentes en une seule.
- Projection à haut contenu
- pathologie numérique
- L'imagerie médicale
Liens externes
- Vaa3D: visualisation et analyse d'images multidimensionnelles hautes performances
- Bioformats Le moteur d'E / S de fichier image qui prend en charge des dizaines de formats