Prédiction génique - Gene prediction

En biologie computationnelle , la prédiction génétique ou la découverte de gènes fait référence au processus d'identification des régions de l'ADN génomique qui codent pour des gènes . Cela inclut les gènes codant pour les protéines ainsi que les gènes ARN , mais peut également inclure la prédiction d'autres éléments fonctionnels tels que les régions régulatrices . La recherche de gènes est l'une des premières et des plus importantes étapes dans la compréhension du génome d'une espèce une fois qu'elle a été séquencée .
À ses débuts, la «découverte de gènes» reposait sur une expérimentation minutieuse sur des cellules et des organismes vivants. L'analyse statistique des taux de recombinaison homologue de plusieurs gènes différents pourrait déterminer leur ordre sur un certain chromosome , et les informations provenant de nombreuses expériences de ce type pourraient être combinées pour créer une carte génétique spécifiant l'emplacement approximatif des gènes connus les uns par rapport aux autres. Aujourd'hui, avec une séquence génomique complète et de puissantes ressources informatiques à la disposition de la communauté de recherche, la recherche de gènes a été redéfinie comme un problème largement informatique.
Il faut distinguer la détermination qu'une séquence est fonctionnelle de la détermination de la fonction du gène ou de son produit. Prédire la fonction d'un gène et confirmer que la prédiction du gène est précise exige toujours une expérimentation in vivo par knock - out de gène et d'autres tests, bien que les frontières de la recherche en bioinformatique rendent de plus en plus possible de prédire la fonction d'un gène en se basant uniquement sur sa séquence.
La prédiction génique est l'une des étapes clés de l'annotation du génome , après l' assemblage des séquences , le filtrage des régions non codantes et le masquage de répétition.
La prédiction génique est étroitement liée au soi-disant «problème de recherche de cible» qui étudie la manière dont les protéines de liaison à l'ADN ( facteurs de transcription ) localisent des sites de liaison spécifiques dans le génome . De nombreux aspects de la prédiction des gènes structuraux sont basés sur la compréhension actuelle des processus biochimiques sous-jacents dans la cellule tels que la transcription génique , la traduction , les interactions protéine-protéine et les processus de régulation , qui font l'objet de recherches actives dans les différents domaines de l' omique tels que la transcriptomique , la protéomique , la métabolomique et plus généralement la génomique structurelle et fonctionnelle .
Méthodes empiriques
Dans les systèmes de recherche de gènes empiriques (similarité, homologie ou factuelle), le génome cible est recherché pour des séquences similaires aux preuves extrinsèques sous la forme des marqueurs de séquence exprimés connus , de l'ARN messager (ARNm), des produits protéiques et des homologues ou séquences orthologues. Étant donné une séquence d'ARNm, il est trivial de dériver une séquence d'ADN génomique unique à partir de laquelle elle devait avoir été transcrite . Étant donné une séquence protéique, une famille de séquences d'ADN codantes possibles peut être dérivée par traduction inverse du code génétique . Une fois que les séquences d'ADN candidates ont été déterminées, il est un problème algorithmique relativement simple de rechercher efficacement dans un génome cible des correspondances, complètes ou partielles, et exactes ou inexactes. Étant donné une séquence, les algorithmes d'alignement local tels que BLAST , FASTA et Smith-Waterman recherchent des régions de similitude entre la séquence cible et d'éventuelles correspondances candidates. Les correspondances peuvent être complètes ou partielles, exactes ou inexactes. Le succès de cette approche est limité par le contenu et la précision de la base de données de séquences.
Un degré élevé de similitude avec un ARN messager ou un produit protéique connu est une preuve solide qu'une région d'un génome cible est un gène codant pour une protéine. Cependant, appliquer cette approche de manière systémique nécessite un séquençage extensif de l'ARNm et des produits protéiques. Non seulement cela est coûteux, mais dans les organismes complexes, seul un sous-ensemble de tous les gènes du génome de l'organisme est exprimé à un moment donné, ce qui signifie que les preuves extrinsèques pour de nombreux gènes ne sont pas facilement accessibles dans une seule culture cellulaire. Ainsi, pour recueillir des preuves extrinsèques pour la plupart ou la totalité des gènes d'un organisme complexe, il faut étudier plusieurs centaines ou milliers de types de cellules , ce qui présente d'autres difficultés. Par exemple, certains gènes humains peuvent être exprimés uniquement au cours du développement sous forme d'embryon ou de fœtus, ce qui peut être difficile à étudier pour des raisons éthiques.
Malgré ces difficultés, de vastes bases de données de transcription et de séquence de protéines ont été générées pour l'homme ainsi que pour d'autres organismes modèles importants en biologie, tels que les souris et la levure. Par exemple, la base de données RefSeq contient des transcriptions et des séquences protéiques de nombreuses espèces différentes, et le système Ensembl cartographie de manière exhaustive ces preuves avec des génomes humains et plusieurs autres. Il est cependant probable que ces bases de données soient à la fois incomplètes et contiennent de petites mais importantes quantités de données erronées.
Les nouvelles technologies de séquençage du transcriptome à haut débit telles que le séquençage ARN-Seq et ChIP ouvrent des opportunités pour incorporer des preuves extrinsèques supplémentaires dans la prédiction et la validation des gènes, et permettent une alternative structurellement riche et plus précise aux méthodes précédentes de mesure de l'expression génique telles que l' étiquette de séquence exprimée ou Puce ADN .
Les principaux défis impliqués dans la prédiction génique impliquent de traiter les erreurs de séquençage dans les données d'ADN brutes, la dépendance à la qualité de l' assemblage des séquences , la gestion des lectures courtes, des mutations de décalage de cadre , des gènes qui se chevauchent et des gènes incomplets.
Chez les procaryotes, il est essentiel de prendre en compte le transfert horizontal de gènes lors de la recherche d'une homologie de séquence génique. Un autre facteur important sous-utilisé dans les outils de détection de gènes actuels est l'existence de groupes de gènes - des opérons (qui sont des unités fonctionnelles d' ADN contenant un groupe de gènes sous le contrôle d'un seul promoteur ) chez les procaryotes et les eucaryotes. Les détecteurs de gènes les plus courants traitent chaque gène de manière isolée, indépendamment des autres, ce qui n'est pas biologiquement précis.
Méthodes ab initio
La prédiction génétique Ab Initio est une méthode intrinsèque basée sur le contenu génétique et la détection du signal. En raison des dépenses et de la difficulté inhérentes à l'obtention de preuves extrinsèques pour de nombreux gènes, il est également nécessaire de recourir à la recherche de gènes ab initio , dans laquelle la séquence d'ADN génomique seule est systématiquement recherchée pour certains signes révélateurs de gènes codant pour des protéines. Ces signes peuvent être globalement classés en tant que signaux , séquences spécifiques qui indiquent la présence d'un gène à proximité, ou contenu , propriétés statistiques de la séquence codant pour la protéine elle-même. La découverte de gènes ab initio pourrait être plus précisément caractérisée comme la prédiction génétique , car des preuves extrinsèques sont généralement nécessaires pour établir de manière concluante qu'un gène putatif est fonctionnel.
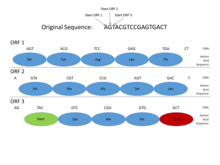
Dans les génomes des procaryotes , les gènes ont des séquences promotrices (signaux) spécifiques et relativement bien comprises , telles que la boîte de Pribnow et les sites de liaison aux facteurs de transcription , qui sont faciles à identifier systématiquement. En outre, la séquence codant pour une protéine se présente sous la forme d'un cadre de lecture ouvert (ORF) contigu , qui est généralement de plusieurs centaines ou milliers de paires de bases de long. Les statistiques des codons stop sont telles que même trouver un cadre de lecture ouvert de cette longueur est un signe assez informatif. (Puisque 3 des 64 codons possibles dans le code génétique sont des codons stop, on s'attendrait à un codon stop environ tous les 20 à 25 codons, ou 60 à 75 paires de bases, dans une séquence aléatoire .) En outre, l'ADN codant pour les protéines a certains périodicités et autres propriétés statistiques faciles à détecter dans une séquence de cette longueur. Ces caractéristiques rendent la recherche de gènes procaryotes relativement simple et des systèmes bien conçus sont capables d'atteindre des niveaux de précision élevés.
La recherche de gènes ab initio chez les eucaryotes , en particulier les organismes complexes comme les humains, est considérablement plus difficile pour plusieurs raisons. Premièrement, le promoteur et les autres signaux de régulation dans ces génomes sont plus complexes et moins bien compris que chez les procaryotes, ce qui les rend plus difficiles à reconnaître de manière fiable. Deux exemples classiques de signaux identifiés par des chercheurs de gènes eucaryotes sont les îlots CpG et les sites de liaison pour une queue poly (A) .
Deuxièmement, les mécanismes d' épissage employés par les cellules eucaryotes signifient qu'une séquence codant pour une protéine particulière dans le génome est divisée en plusieurs parties ( exons ), séparées par des séquences non codantes ( introns ). (Les sites d'épissage sont eux-mêmes un autre signal que les chercheurs de gènes eucaryotes sont souvent conçus pour identifier.) Un gène codant une protéine typique chez l'homme peut être divisé en une douzaine d'exons, chacun de moins de deux cents paires de bases de longueur, et certains aussi courts que vingt à trente. Il est donc beaucoup plus difficile de détecter les périodicités et autres propriétés de contenu connues de l'ADN codant pour les protéines chez les eucaryotes.
Les chercheurs de gènes avancés pour les génomes procaryotes et eucaryotes utilisent généralement des modèles probabilistes complexes , tels que les modèles de Markov cachés (HMM) pour combiner des informations provenant d'une variété de mesures de signaux et de contenus différents. Le système GLIMMER est un chercheur de gènes largement utilisé et très précis pour les procaryotes. GeneMark est une autre approche populaire. Les chercheurs de gènes eucaryotes ab initio , par comparaison, n'ont obtenu qu'un succès limité; des exemples notables sont les programmes GENSCAN et geneid . Le chercheur de gène SNAP est basé sur HMM comme Genscan, et tente d'être plus adaptable à différents organismes, en résolvant les problèmes liés à l'utilisation d'un chercheur de gène sur une séquence du génome contre laquelle il n'a pas été formé. Quelques approches récentes telles que mSplicer, CONTRAST ou mGene utilisent également des techniques d' apprentissage automatique telles que des machines à vecteurs de support pour une prédiction génique réussie. Ils construisent un modèle discriminant à l' aide de machines vectorielles de support de Markov cachées ou de champs aléatoires conditionnels pour apprendre une fonction de score de prédiction génique précise.
Les méthodes Ab Initio ont été comparées, certaines avec une sensibilité approchant les 100%, mais à mesure que la sensibilité augmente, la précision souffre en raison de l'augmentation des faux positifs .
Autres signaux
Parmi les signaux dérivés utilisés pour la prédiction se trouvent les statistiques résultant des statistiques de sous-séquence comme les statistiques k-mer , Isochore (génétique) ou composition GC de domaine composition / uniformité / entropie, séquence et longueur de trame, Intron / Exon / Donor / Acceptor / Promoter et le vocabulaire du site de liaison ribosomique , la dimension fractale , la transformée de Fourier d'un ADN codé en pseudo-nombre, les paramètres de la courbe en Z et certaines caractéristiques de l'analyse.
Il a été suggéré que des signaux autres que ceux directement détectables dans les séquences peuvent améliorer la prédiction génique. Par exemple, le rôle de la structure secondaire dans l'identification des motifs régulateurs a été rapporté. De plus, il a été suggéré que la prédiction de la structure secondaire de l'ARN aide à la prédiction du site d'épissage.
Les réseaux de neurones
Les réseaux de neurones artificiels sont des modèles informatiques qui excellent dans l'apprentissage automatique et la reconnaissance de formes . Les réseaux de neurones doivent être formés avec des exemples de données avant de pouvoir généraliser pour des données expérimentales, et testés par rapport à des données de référence. Les réseaux de neurones sont capables de proposer des solutions approximatives à des problèmes difficiles à résoudre de manière algorithmique, à condition qu'il y ait suffisamment de données d'apprentissage. Lorsqu'ils sont appliqués à la prédiction génique, les réseaux neuronaux peuvent être utilisés avec d'autres méthodes ab initio pour prédire ou identifier des caractéristiques biologiques telles que les sites d'épissage. Une approche consiste à utiliser une fenêtre glissante, qui parcourt les données de séquence de manière chevauchante. La sortie à chaque position est un score basé sur le fait que le réseau pense que la fenêtre contient un site d'épissure donneur ou un site d'épissure accepteur. Les fenêtres plus grandes offrent plus de précision, mais nécessitent également plus de puissance de calcul. Un réseau neuronal est un exemple de capteur de signal car son objectif est d'identifier un site fonctionnel dans le génome.
Approches combinées
Des programmes tels que Maker combinent des approches extrinsèques et ab initio en cartographiant les données protéiques et EST sur le génome pour valider les prédictions ab initio . Augustus , qui peut être utilisé dans le cadre du pipeline Maker, peut également incorporer des indices sous la forme d'alignements EST ou de profils de protéines pour augmenter la précision de la prédiction des gènes.
Approches génomiques comparatives
Comme les génomes entiers de nombreuses espèces différentes sont séquencés, une approche prometteuse dans la recherche actuelle sur la recherche de gènes est une approche génomique comparative .
Ceci est basé sur le principe que les forces de la sélection naturelle font que les gènes et autres éléments fonctionnels subissent une mutation à un rythme plus lent que le reste du génome, car les mutations dans les éléments fonctionnels sont plus susceptibles d'avoir un impact négatif sur l'organisme que les mutations ailleurs. Les gènes peuvent ainsi être détectés en comparant les génomes d'espèces apparentées pour détecter cette pression évolutive pour la conservation. Cette approche a d'abord été appliquée aux génomes de la souris et de l'homme, en utilisant des programmes tels que SLAM, SGP et TWINSCAN / N-SCAN et CONTRAST.
Plusieurs informateurs
TWINSCAN a examiné uniquement la synthèse homme-souris pour rechercher des gènes orthologues. Des programmes tels que N-SCAN et CONTRAST ont permis l'incorporation d'alignements à partir de plusieurs organismes, ou dans le cas de N-SCAN, un seul organisme alternatif de la cible. L'utilisation de plusieurs informateurs peut conduire à des améliorations significatives de l'exactitude.
CONTRAST est composé de deux éléments. Le premier est un classificateur plus petit, identifiant les sites d'épissage donneur et les sites d'épissage accepteur ainsi que les codons de démarrage et d'arrêt. Le deuxième élément consiste à construire un modèle complet à l'aide de l'apprentissage automatique. Diviser le problème en deux signifie que des ensembles de données ciblés plus petits peuvent être utilisés pour former les classificateurs, et ce classificateur peut fonctionner indépendamment et être formé avec des fenêtres plus petites. Le modèle complet peut utiliser le classificateur indépendant et ne pas avoir à perdre de temps de calcul ou de complexité du modèle à reclassifier les limites intron-exon. L'article dans lequel CONTRAST est introduit propose que leur méthode (et celles de TWINSCAN, etc.) soit classée comme assemblage de gènes de novo , en utilisant des génomes alternatifs, et en l'identifiant comme distinct de ab initio , qui utilise un génome «informateur» cible.
La recherche comparative de gènes peut également être utilisée pour projeter des annotations de haute qualité d'un génome à un autre. Les exemples notables incluent Projector, GeneWise, GeneMapper et GeMoMa. Ces techniques jouent désormais un rôle central dans l'annotation de tous les génomes.
Prédiction pseudogène
Les pseudogènes sont des parents proches des gènes, partageant une homologie de séquence très élevée, mais étant incapables de coder pour le même produit protéique . Alors qu'ils étaient autrefois relégués comme sous-produits du séquençage des gènes , de plus en plus, à mesure que les rôles de régulation sont découverts, ils deviennent des cibles prédictives à part entière. La prédiction de pseudogène utilise la similarité de séquence existante et les méthodes ab initio, tout en ajoutant un filtrage supplémentaire et des méthodes d'identification des caractéristiques du pseudogène.
Les méthodes de similarité de séquence peuvent être personnalisées pour la prédiction de pseudogènes en utilisant un filtrage supplémentaire pour trouver des pseudogènes candidats. Cela pourrait utiliser la détection de désactivation, qui recherche des mutations absurdes ou à décalage de cadre qui tronqueraient ou réduiraient une séquence codante par ailleurs fonctionnelle. De plus, la traduction de l'ADN en séquences de protéines peut être plus efficace qu'une simple homologie d'ADN.
Les capteurs de contenu peuvent être filtrés en fonction des différences de propriétés statistiques entre les pseudogènes et les gènes, telles qu'un nombre réduit d'îlots CpG dans les pseudogènes, ou les différences de contenu en GC entre les pseudogènes et leurs voisins. Les capteurs de signal peuvent également être adaptés aux pseudogènes, à la recherche de l'absence d'introns ou de queues de polyadénine.
Prédiction des gènes métagénomiques
La métagénomique est l'étude du matériel génétique récupéré de l'environnement, résultant en des informations de séquence à partir d'un pool d'organismes. La prédiction des gènes est utile pour la métagénomique comparative .
Les outils de métagénomique entrent également dans les catégories de base d'utilisation des approches de similarité de séquence (MEGAN4) et des techniques ab initio (GLIMMER-MG).
Glimmer-MG est une extension de GLIMMER qui repose principalement sur une approche ab initio pour la recherche de gènes et en utilisant des ensembles de formation provenant d'organismes apparentés. La stratégie de prédiction est complétée par la classification et le regroupement des ensembles de données génétiques avant d'appliquer des méthodes ab initio de prédiction génique. Les données sont regroupées par espèce. Cette méthode de classification tire parti des techniques de la classification phylogénétique métagénomique. Un exemple de logiciel à cet effet est Phymm, qui utilise des modèles markov interpolés, et PhymmBL, qui intègre BLAST dans les routines de classification.
MEGAN4 utilise une approche de similarité de séquence, utilisant l'alignement local contre des bases de données de séquences connues, mais tente également de classer en utilisant des informations supplémentaires sur les rôles fonctionnels, les voies biologiques et les enzymes. Comme dans le cas de la prédiction génique d'un seul organisme, les approches de similarité de séquence sont limitées par la taille de la base de données.
FragGeneScan et MetaGeneAnnotator sont des programmes de prédiction génique populaires basés sur le modèle de Markov caché . Ces prédicteurs tiennent compte des erreurs de séquençage, des gènes partiels et fonctionnent pour de courtes lectures.
Un autre outil rapide et précis pour la prédiction génétique dans les métagénomes est MetaGeneMark. Cet outil est utilisé par le DOE Joint Genome Institute pour annoter IMG / M, la plus grande collection de métagénomes à ce jour.
Voir également
- Liste des logiciels de prédiction génique
- Extraction de séquences
- Prédiction de la fonction des protéines
- Empreinte phylogénétique
- Similitude de séquence (homologie)
Liens externes
- Auguste
- FGENESH
- GeMoMa - Prédiction génétique basée sur l'homologie basée sur la conservation de la position des acides aminés et des introns ainsi que sur les données RNA-Seq
- geneid , SGP2
- Glimmer , GlimmerHMM
- GenomeThreader
- ChemGenome
- GeneMark
- Gismo
- mGene
- StarORF - Un outil multi-plateforme et web pour prédire les ORF et obtenir la séquence du complément inverse
- Maker - Un pipeline d'annotation de génome portable et facilement configurable
