Projet sur le génome du chimpanzé - Chimpanzee genome project
Le Chimpanzee Genome Project était un effort pour déterminer la séquence d' ADN du génome du chimpanzé . Le séquençage a commencé en 2005 et en 2013, vingt-quatre chimpanzés individuels avaient été séquencés. Ce projet a été intégré au Great Ape Genome Project .

En 2013, des séquences à haute résolution ont été publiées pour chacune des quatre sous-espèces de chimpanzés reconnues : Central chimpanzee , Pan troglodytes troglodytes , 10 séquences ; Chimpanzé occidental , Pan troglodytes verus , 6 séquences; Chimpanzé du Nigeria-Cameroun , Pan troglodytes ellioti , 4 séquences ; et le chimpanzé de l'Est , Pan troglodytes schweinfurthii , 4 séquences. Ils ont tous été séquencés à une couverture moyenne de 25 fois par individu.
La recherche a montré une diversité génomique considérable chez les chimpanzés avec de nombreux traits spécifiques à la population. Les chimpanzés du centre conservent la plus grande diversité dans la lignée des chimpanzés, tandis que les autres sous-espèces présentent des signes de goulot d'étranglement de la population .
Fond
Les chromosomes humains et chimpanzés sont très similaires. La principale différence est que les humains ont une paire de chromosomes de moins que les autres grands singes . Les humains ont 23 paires de chromosomes et les autres grands singes ont 24 paires de chromosomes. Dans la lignée évolutive humaine, deux chromosomes ancestraux de singe ont fusionné au niveau de leurs télomères , produisant le chromosome humain 2 . Il existe neuf autres différences chromosomiques majeures entre les chimpanzés et les humains : des inversions de segments chromosomiques sur les chromosomes humains 1 , 4 , 5 , 9 , 12 , 15 , 16 , 17 et 18 . Après l'achèvement du projet sur le génome humain , un projet commun sur le génome des chimpanzés a été lancé. En décembre 2003, une analyse préliminaire de 7600 gènes partagés entre les deux génomes a confirmé que certains gènes comme le facteur de transcription forkhead-box P2 , qui est impliqué dans le développement de la parole, sont différents dans la lignée humaine. Plusieurs gènes impliqués dans l'audition se sont également avérés avoir changé au cours de l'évolution humaine, suggérant une sélection impliquant un comportement lié au langage humain . Les différences entre les humains individuels et les chimpanzés communs sont estimées à environ 10 fois la différence typique entre les paires d'humains.
Une autre étude a montré que les modèles de méthylation de l'ADN, qui sont un mécanisme de régulation connu de l'expression des gènes, diffèrent dans le cortex préfrontal des humains par rapport aux chimpanzés, et ont impliqué cette différence dans la divergence évolutive des deux espèces.

Projet de séquence du génome du chimpanzé commun
Une analyse de la séquence du génome chimpanzé a été publiée dans Nature le 1er Septembre 2005, dans un article produit par le Chimpanzé séquençage et analyse Consortium , un groupe de scientifiques qui est soutenu en partie par l' Institut national de recherche sur le génome humain , l' un des National Instituts de Santé . L'article marquait l'achèvement du projet de séquence du génome.
Il existe maintenant une base de données contenant les différences génétiques entre les gènes humains et chimpanzés, avec environ trente-cinq millions de changements de nucléotides simples , cinq millions d' événements d' insertion/délétion et divers réarrangements chromosomiques . Les duplications de gènes expliquent la plupart des différences de séquences entre les humains et les chimpanzés. Les substitutions de paires de bases uniques représentent environ la moitié des changements génétiques que la duplication de gènes.
Humain typique et chimpanzés homologues de protéines diffèrent par seulement une moyenne de deux acides aminés . Environ 30 pour cent de toutes les protéines humaines sont identiques en séquence à la protéine de chimpanzé correspondante. Comme mentionné ci-dessus, les duplications de gènes sont une source majeure de différences entre le matériel génétique humain et chimpanzé, avec environ 2,7 pour cent du génome représentant maintenant des différences ayant été produites par des duplications ou des suppressions de gènes pendant environ 6 millions d'années depuis que les humains et les chimpanzés ont divergé de leur ancêtre évolutif. La variation comparable au sein des populations humaines est de 0,5 pour cent.
Environ 600 gènes ont été identifiés qui pourraient avoir subi une forte sélection positive dans les lignées humaines et chimpanzées ; nombre de ces gènes sont impliqués dans la défense du système immunitaire contre les maladies microbiennes (exemple : la granulysine est protectrice contre Mycobacterium tuberculosis ) ou sont des récepteurs ciblés de micro-organismes pathogènes (exemple : Glycophorine C et Plasmodium falciparum ). En comparant les gènes humains et de chimpanzé aux gènes d'autres mammifères, il a été découvert que les gènes codant pour des facteurs de transcription , tels que forkhead-box P2 ( FOXP2 ), ont souvent évolué plus rapidement chez l'humain que chez le chimpanzé ; des changements relativement faibles dans ces gènes peuvent expliquer les différences morphologiques entre les humains et les chimpanzés. Un ensemble de 348 gènes de facteurs de transcription code pour des protéines avec une moyenne d'environ 50 pour cent plus de changements d'acides aminés dans la lignée humaine que dans la lignée des chimpanzés.
Six régions chromosomiques humaines ont été trouvées qui pourraient avoir été soumises à une sélection particulièrement forte et coordonnée au cours des 250 000 dernières années. Ces régions contiennent au moins un allèle marqueur qui semble unique à la lignée humaine, tandis que l'ensemble de la région chromosomique présente une variation génétique inférieure à la normale. Ce schéma suggère qu'un ou quelques gènes fortement sélectionnés dans la région chromosomique peuvent avoir empêché l'accumulation aléatoire de changements neutres dans d'autres gènes voisins. Une de ces régions sur le chromosome 7 contient le gène FOXP2 (mentionné ci-dessus) et cette région comprend également le gène régulateur de la conductance transmembranaire de la mucoviscidose (CFTR), qui est important pour le transport des ions dans les tissus tels que l'épithélium sécrétant du sel des glandes sudoripares. Des mutations humaines dans le gène CFTR pourraient être sélectionnées pour survivre au choléra .
Une autre région de ce type sur le chromosome 4 peut contenir des éléments régulant l'expression d'un gène de protocadhérine voisin qui peut être important pour le développement et le fonctionnement du cerveau . Bien que les modifications de l'expression des gènes exprimés dans le cerveau aient tendance à être moindres que pour d'autres organes (comme le foie) en moyenne, les modifications de l'expression des gènes dans le cerveau ont été plus spectaculaires dans la lignée humaine que dans la lignée des chimpanzés. Ceci est cohérent avec la divergence dramatique du modèle unique de développement du cerveau humain observé dans la lignée humaine par rapport au modèle ancestral des grands singes. Le groupe de gènes protocadhérine-bêta sur le chromosome 5 montre également des preuves d'une sélection positive possible.
Les résultats des analyses du génome humain et chimpanzé devraient aider à comprendre certaines maladies humaines. Les humains semblent avoir perdu un gène fonctionnel de la caspase 12 , qui chez d'autres primates code pour une enzyme qui pourrait protéger contre la maladie d'Alzheimer .
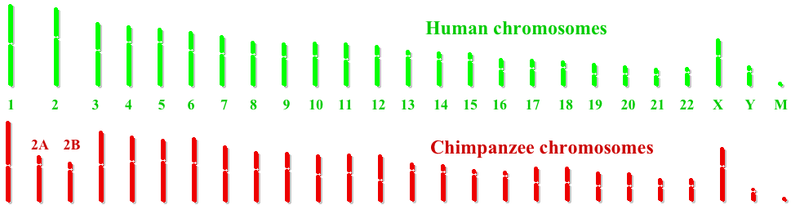
Gènes du site de fusion du chromosome 2
Les résultats du projet sur le génome du chimpanzé suggèrent que lorsque les chromosomes ancestraux 2A et 2B ont fusionné pour produire le chromosome humain 2, aucun gène n'a été perdu des extrémités fusionnées de 2A et 2B. Au site de fusion, il y a environ 150 000 paires de bases de séquence non trouvées dans les chromosomes 2A et 2B du chimpanzé. Des copies liées supplémentaires des gènes PGML/FOXD/CBWD existent ailleurs dans le génome humain, en particulier près de l'extrémité p du chromosome 9 . Cela suggère qu'une copie de ces gènes peut avoir été ajoutée à la fin de l'ancêtre 2A ou 2B avant l'événement de fusion. Il reste à déterminer si ces gènes insérés confèrent un avantage sélectif.
- PGM5P4 . Le pseudogène de la phosphoglucomutase du chromosome 2 humain. Ce gène est incomplet et ne produit pas de transcrit fonctionnel.
- FOXD4L1 . Le gène de type Forkhead box D4 est un exemple de gène sans intron. La fonction de ce gène n'est pas connue, mais il pourrait coder pour une protéine de contrôle de la transcription.
- CBWD2 . La cobalamine synthétase est une enzyme bactérienne qui fabrique la vitamine B 12 . Dans un passé lointain, un ancêtre commun aux souris et aux singes incorporait une copie d'un gène de la cobalamine synthétase (voir : Transfert horizontal de gènes ). Les humains ont la particularité de posséder plusieurs copies de gènes de type cobalamine synthétase, dont celui sur le chromosome 2. Il reste à déterminer quelle est la fonction de ces gènes humains de type cobalamine synthétase. Si ces gènes sont impliqués dansvitamine B 12 métabolisme, cela pourrait être utile àévolution humaine. Un changement majeur dans le développement humain est une croissance cérébrale post-natale plus importante que celle observée chez les autres singes. La vitamine B 12 est importante pour le développement du cerveau, et une carence en vitamine B 12 pendant le développement du cerveau entraîne de graves anomalies neurologiques chez les enfants humains.
- WASH2P . Plusieurs transcrits de fonction inconnue correspondant à cette région ont été isolés. Cette région est également présente dans la région terminale étroitement liée du chromosome 9p qui contient des copies des gènes PGML/FOXD/CBWD.
- RPL23AP7 . De nombreux pseudogènes de la protéine ribosomique L23a sont dispersés dans le génome humain.
Voir également
Lectures complémentaires
- Suntsova, MV ; Buzdin, AA (2020-09-10). "Différences entre les génomes humains et chimpanzés et leurs implications dans l'expression des gènes, les fonctions des protéines et les propriétés biochimiques des deux espèces" . BMC Génomique . 21 (535) : 535. doi : 10.1186/s12864-020-06962-8 . PMC 7488140 . PMID 32912141 .
Les références
-
^ un b Prado-Martinez, J.; et al. (2013). "Diversité génétique et histoire de la population des grands singes" . Nature . 499 (7459) : 471-475. Bibcode : 2013Natur.499..471P . doi : 10.1038/nature12228 . PMC 3822165 . PMID 23823723 .

- ^ Bosquets, Colin P. (2001). Taxonomie des primates . Washington, DC : Smithsonian Institution Press. p. 303-307. ISBN 978-1-56098-872-4.
- ^ Hof, J.; Sommer, V. (2010). Des singes comme nous : portraits d'une parenté . Mannheim : Panorama. p. 114. ISBN 978-3-89823-435-1.
- ^ de Manuel, M.; et al. (2016). "La diversité génomique des chimpanzés révèle un ancien mélange avec les bonobos" . Sciences . 354 (6311) : 477–48. Bibcode : 2016Sci ... 354..477D . doi : 10.1126/science.aag2602 . PMC 5546212 . PMID 27789843 .
- ^ De Grouchy J (août 1987). "Phylogénies chromosomiques de l'homme, des grands singes et des singes du Vieux Monde". Génétique . 73 (1-2) : 37-52. doi : 10.1007/bf00057436 . PMID 3333352 . S2CID 1098866 .
-
^ a b Séquençage de chimpanzés; Consortium d'analyse (2005). « Séquence initiale du génome du chimpanzé et comparaison avec le génome humain » (PDF) . Nature . 437 (7055) : 69-87. Code Bib : 2005Natur.437...69. . doi : 10.1038/nature04072 . PMID 16136131 .
-
^ Zeng, J.; Konopa, G.; Chasse, BG ; Preuss, TM ; Geschwind, D.; Yi, SV (2012). "Les cartes divergentes de la méthylation du génome entier des cerveaux humains et de chimpanzés révèlent la base épigénétique de l'évolution de la réglementation humaine" . Le Journal américain de génétique humaine . 91 (3) : 455-465. doi : 10.1016/j.ajhg.2012.07.024 . PMC 3511995 . PMID 22922032 .

- ^ McConkey EH (2004). « La numérotation orthologue des grands singes et des chromosomes humains est essentielle pour la génomique comparative » . Cytogenète. Génome Res . 105 (1) : 157–8. doi : 10.1159/000078022 . PMID 15218271 . S2CID 11571357 .
- ^ Springer MS, Murphy WJ, Eizirik E, O'Brien SJ (février 2003). "La diversification placentaire des mammifères et la limite Crétacé-Tertiaire" . Proc. Natl. Acad. Sci. États-Unis . 100 (3) : 1056–61. Bibcode : 2003PNAS..100.1056S . doi : 10.1073/pnas.0334222100 . PMC 298725 . PMID 12552136 .
- ^ "Base de données du génome des chimpanzés (Genome Data Viewer Pan troglodytes (chimpanzé))" .
- ^ Caswell JL, Mallick S, Richter DJ, Neubauer J, Schirmer C, Gnerre S, Reich D (avril 2008). "Analyse de l'histoire des chimpanzés basée sur les alignements de séquences de génomes" . PLOS Genet . 4 (4) : e1000057. doi : 10.1371/journal.pgen.1000057 . PMC 2278377 . PMID 18421364 .
- ^ Cheng Z, Ventura M, She X, Khaitovich P, Graves T, Osoegawa K, et al. (septembre 2005). « Une comparaison à l'échelle du génome des duplications segmentaires récentes de chimpanzés et humains ». Nature . 437 (7055) : 88-93. Code Bib : 2005Natur.437...88C . doi : 10.1038/nature04000 . PMID 16136132 . S2CID 4420359 .
- ^ Stenger S, Hanson DA, Teitelbaum R, Dewan P, Niazi KR, Froelich CJ, et al. (octobre 1998). « Une activité antimicrobienne des cellules T cytolytiques médiée par la granulysine ». Sciences . 282 (5386) : 121-5. Bibcode : 1998Sci ... 282..121S . doi : 10.1126/science.282.5386.121 . PMID 9756476 .
- ^ Goodman BE, Percy WH (juin 2005). « CFTR dans la mucoviscidose et le choléra : du transport membranaire à la pratique clinique ». Adv Physiol Educ . 29 (2) : 75-82. doi : 10.1152/advan.00035.2004 . PMID 15905150 .
- ^ Khaitovich P, Hellmann I, Enard W, Nowick K, Leinweber M, Franz H, Weiss G, Lachmann M, Pääbo S (septembre 2005). « Modèles parallèles d'évolution dans les génomes et les transcriptomes des humains et des chimpanzés ». Sciences . 309 (5742) : 1850–4. Bibcode : 2005Sci ... 309.1850K . doi : 10.1126/science.1108296 . PMID 16141373 . S2CID 16674740 .
- ^ Miki R, Hattori K, Taguchi Y, Tada MN, Isosaka T, Hidaka Y, Hirabayashi T, Hashimoto R, Fukuzako H, Yagi T (avril 2005). « Identification et caractérisation du codage des polymorphismes mononucléotidiques au sein des groupes de gènes humains protocadhérine-alpha et -bêta ». Gène . 349 : 1-14. doi : 10.1016/j.gene.2004.11.044 . PMID 15777644 .
- ^ Fan Y, Newman T, Linardopoulou E, Trask BJ (novembre 2002). "Contenu génique et fonction du site de fusion chromosomique ancestral dans le chromosome humain 2q13-2q14.1 et les régions paralogues" . Génome Res . 12 (11) : 1663-1672. doi : 10.1101/gr.338402 . PMC 187549 . PMID 12421752 .

