Nucléoïde - Nucleoid
Le nucléoïde (c'est-à-dire semblable au noyau ) est une région de forme irrégulière à l'intérieur de la cellule procaryote qui contient tout ou la majeure partie du matériel génétique . Le chromosome d'un procaryote est circulaire et sa longueur est très grande par rapport aux dimensions de la cellule nécessitant qu'il soit compacté pour s'adapter. Contrairement au noyau d'une cellule eucaryote , il n'est pas entouré d'une membrane nucléaire . Au lieu de cela, le nucléoïde se forme par condensation et arrangement fonctionnel à l'aide de protéines architecturales chromosomiques et de molécules d' ARN ainsi que par superenroulement d'ADN . La longueur d'un génome est très variable (généralement au moins quelques millions de paires de bases) et une cellule peut en contenir plusieurs copies.
Il n'y a pas encore de structure à haute résolution connue d'un nucléoïde bactérien, mais des caractéristiques clés ont été recherchées chez Escherichia coli en tant qu'organisme modèle . Chez E. coli , l'ADN chromosomique est en moyenne surenroulé négativement et replié en boucles plectonémiques , qui sont confinées à différentes régions physiques, et diffusent rarement les unes dans les autres. Ces boucles s'organisent spatialement en régions de la taille d'une mégabase appelées macrodomaines, au sein desquelles les sites d'ADN interagissent fréquemment, mais entre lesquels les interactions sont rares. L'ADN condensé et organisé spatialement forme un ellipsoïde hélicoïdal qui est radialement confiné dans la cellule. La structure 3D de l'ADN dans le nucléoïde semble varier selon les conditions et est liée à l'expression des gènes, de sorte que l'architecture nucléoïde et la transcription des gènes sont étroitement interdépendantes, s'influencent mutuellement.

Fond
Dans de nombreuses bactéries, le chromosome est une seule molécule d'ADN double brin fermée de manière covalente (circulaire) qui code l'information génétique sous une forme haploïde . La taille de l'ADN varie de 500 000 à plusieurs millions de paires de bases (pb) codant de 500 à plusieurs milliers de gènes selon les organismes. L'ADN chromosomique est présent dans les cellules sous une forme organisée et très compacte appelée nucléoïde (c'est -à-dire semblable à un noyau ), qui n'est pas enfermée dans une membrane nucléaire comme dans les cellules eucaryotes. Le nucléoïde isolé contient 80 % d'ADN, 10 % de protéines et 10 % d'ARN en poids.
La bactérie à Gram négatif Escherichia coli est un système modèle pour la recherche nucléoïde sur la façon dont l'ADN chromosomique devient le nucléoïde, les facteurs qui y sont impliqués, ce que l'on sait de sa structure et comment certains aspects structurels de l'ADN influencent l'expression des gènes .
Il y a deux aspects essentiels de la formation nucléoïde ; condensation d'un grand ADN dans un petit espace cellulaire et organisation fonctionnelle de l'ADN sous une forme tridimensionnelle. Le chromosome circulaire haploïde chez E. coli se compose de ~ 4,6 x 10 6 pb. Si l'ADN est relaxé sous la forme B , il aurait une circonférence d'environ 1,5 millimètres (0,332 nm x 4,6 x 10 6 ). Cependant, une grosse molécule d'ADN telle que l' ADN chromosomique d' E. coli ne reste pas une molécule rigide et droite dans une suspension. Le mouvement brownien générera une courbure et des courbures dans l'ADN. La longueur maximale jusqu'à laquelle un ADN à double hélice reste droit en résistant à la flexion imposée par le mouvement brownien est d'environ 50 nm ou 150 pb, appelée longueur de persistance . Ainsi, l'ADN pur se condense sensiblement sans aucun facteur supplémentaire ; à l'équilibre thermique, il prend une forme de bobine aléatoire . La bobine aléatoire d' ADN chromosomique d' E. coli occuperait un volume (4/3 π r 3 ) de ~ 523 µm 3 , calculé à partir du rayon de giration ( R g = (√N a)/√6) où a est le Longueur de Kuhn (2 x longueur de persistance), et N est le nombre de segments de longueur de Kuhn dans l'ADN (longueur totale de l'ADN divisée par a ). Bien que l'ADN soit déjà condensé sous forme de bobine aléatoire, il ne peut toujours pas assumer le volume du nucléoïde qui est inférieur à un micron. Ainsi, la propriété inhérente de l'ADN n'est pas suffisante : des facteurs supplémentaires doivent aider à condenser davantage l'ADN de l'ordre de ~10 3 (volume de la bobine aléatoire divisé par le volume nucléoïde). Le deuxième aspect essentiel de la formation des nucléoïdes est l'arrangement fonctionnel de l'ADN. L'ADN chromosomique n'est pas seulement condensé mais également organisé fonctionnellement d'une manière compatible avec les processus de transaction de l'ADN tels que la réplication , la recombinaison , la ségrégation et la transcription . Près de cinq décennies de recherche, commencées en 1971, ont montré que la forme finale du nucléoïde résulte d'une organisation hiérarchique de l'ADN. À la plus petite échelle (1 kb ou moins), les protéines architecturales de l'ADN associées aux nucléoïdes condensent et organisent l'ADN en pliant, en boucle, en pontant ou en enveloppant l'ADN. À plus grande échelle (10 kb ou plus), l'ADN forme des boucles plétonémiques, une forme tressée d'ADN induite par superenroulement. À l'échelle de la mégabase, les boucles plétonémiques fusionnent en six domaines organisés spatialement (macrodomaines), qui sont définis par des interactions physiques plus fréquentes entre les sites d'ADN au sein du même macrodomaine qu'entre différents macrodomaines. Les connexions ADN-ADN à longue et courte portée formées à l'intérieur et entre les macrodomaines contribuent à la condensation et à l'organisation fonctionnelle. Enfin, le nucléoïde est un ellipsoïde hélicoïdal avec des régions d'ADN hautement condensé sur l'axe longitudinal.
Condensation et organisation
Protéines associées aux nucléoïdes (NAP)
Chez les eucaryotes, l'ADN génomique est condensé sous la forme d'un réseau répétitif de particules ADN-protéines appelées nucléosomes .
Un nucléosome est constitué d'environ 146 pb d'ADN enroulé autour d'un complexe octamère des protéines histones . Bien que les bactéries n'aient pas d'histones, elles possèdent un groupe de protéines de liaison à l'ADN appelées protéines associées aux nucléoïdes (NAP) qui sont fonctionnellement analogues aux histones au sens large. Les NAP sont très abondantes et constituent une proportion importante de la composante protéique du nucléoïde.
Une caractéristique distinctive des NAP est leur capacité à se lier à l'ADN à la fois d'une manière spécifique (spécifique à la séquence ou à la structure) et non spécifique à la séquence. En conséquence, les NAP sont des protéines à double fonction. La liaison spécifique des NAP est principalement impliquée dans la transcription spécifique du gène , la réplication de l' ADN , la recombinaison et la réparation . Au sommet de leur abondance, le nombre de molécules de nombreuses NAP est supérieur de plusieurs ordres de grandeur au nombre de sites de liaison spécifiques dans le génome. Par conséquent, on pense que les NAP se lient à l'ADN chromosomique principalement dans le mode non spécifique de séquence et c'est ce mode qui est crucial pour la compaction des chromosomes. Il est à noter que ce que l'on appelle la liaison spécifique non séquentielle d'un NAP peut ne pas être complètement aléatoire. Il pourrait y avoir une faible spécificité de séquence et/ou une spécificité structurelle due à une conformation d'ADN dépendante de la séquence ou à une conformation d'ADN créée par d'autres NAP.
Bien que les mécanismes moléculaires de la façon dont les NAP condensent l'ADN in vivo ne soient pas bien compris, sur la base des études in vitro approfondies , il semble que les NAP participent au compactage des chromosomes via les mécanismes suivants : les NAP induisent et stabilisent les courbures de l'ADN, aidant ainsi à la condensation de l'ADN en réduisant la longueur de persistance. Les NAP condensent l'ADN en pontant, en enveloppant et en groupant qui pourraient se produire entre des segments d'ADN proches ou des segments d'ADN distants du chromosome. Un autre mécanisme par lequel les NAP participent à la compaction des chromosomes consiste à contraindre les superbobines négatives dans l'ADN, contribuant ainsi à l'organisation topologique du chromosome.
Il existe au moins 12 NAP identifiées dans E. coli, dont les plus étudiées sont HU, IHF, H-NS et Fis. Leur abondance et leurs propriétés de liaison à l'ADN et leur effet sur la condensation et l'organisation de l'ADN sont résumés dans les tableaux ci-dessous.
| Protéine | Masse moléculaire (kDa) | Unité fonctionnelle native | Abondance 1 en phase de croissance | Abondance 1 en phase stationnaire |
|---|---|---|---|---|
| HUα et HUβ | ~ 9 | Homo- et hétéro-dimères | 55 000 (23) | 30 000 (12,5) |
| IHFα et IHFβ | ~ 11 | Hétérodimère | 12 000 (5) | 55 000 (23) |
| H-NS | ~ 15 | Homodimère | 20 000 (8) | 15 000 (6) |
| Fis | ~ 11 | Homodimère | 60 000 (25) | Indétectable |
| Dps | ~ 19 | Dodécamère | 6 000 (0,4) | 180 000 (12,5) |
1 Les données d' abondance (molécules/cellule) ont été tirées de ; Le nombre entre parenthèses est la concentration micromolaire calculée selon la formule suivante : (nombre d'unités fonctionnelles natives/nombre d'Avogadro) x (1/volume cellulaire en litre) x 10 3 . Le volume cellulaire en litre (2 x 10 -15 ) a été déterminé en supposant que le volume de la cellule d' E. coli était de 2 m 3 .
| Protéine | Motif de reliure | Affinité de liaison à l'ADN spécifique 1 | Affinité de liaison à l'ADN aléatoire 1 |
|---|---|---|---|
| HU | Un motif structurel défini par des courbures et des plis de l'ADN | 7,5 x 10 -9 | 4,0 x 10 -7 |
| H-NS | WATCAANNNNTTR | 1,5 x 10 -9 | 1,7 x 10 -6 |
| IHF | TCGATAAATT | 10-15 x 10 -9 | 6 x 10 -8 |
| Fis | GNTYAAAWTTTRANC | 0,2-1,0 x 10 -9 | >8,0 x 10 -6 |
| Dps | ND | ND | 1,65 x 10 -7 |
| MatP | GTGACRNYGTCAC | 8,0 x 10 -9 | ND |
| MukBEF | ND | ND | ND |
1 L' affinité de liaison fait référence à la constante de dissociation à l'équilibre (Kd) en unités molaires (M). ND = non déterminé
HU
La protéine de type histone de la souche U93 (HU) d' E. coli est une protéine conservée au cours de l'évolution chez les bactéries. HU existe dans E. coli sous forme d'homo- et d'hétérodimères de deux sous-unités HUα et HUβ partageant 69 % d'identité en acides aminés. Bien qu'on l'appelle une protéine de type histone, les proches parents fonctionnels de l'HU chez les eucaryotes sont des protéines du groupe à haute mobilité (HMG), et non des histones. HU est une protéine de liaison à l'ADN non spécifique à une séquence. Il se lie avec une faible affinité à tout ADN linéaire. Cependant, il se lie préférentiellement avec une haute affinité à un ADN structurellement déformé. Des exemples de substrats d'ADN déformés comprennent l' ADN cruciforme, l'ADN bombé, l'ADNdb contenant une cassure simple brin telle que des entailles , des espaces ou des fourches . De plus, HU se lie spécifiquement et stabilise une boucle d'ADN à médiation protéique. Dans le mode de liaison à l'ADN structurellement spécifique, HU reconnaît un motif structurel commun défini par des courbures ou des plis créés par la distorsion, alors qu'il se lie à un ADN linéaire en verrouillant le squelette phosphate. Alors que la liaison structurellement spécifique de haute affinité est requise pour les fonctions spécialisées de l'HU telles que la recombinaison spécifique au site , la réparation de l' ADN , l' initiation de la réplication de l'ADN et la régulation des gènes, il semble que la liaison générale de faible affinité soit impliquée dans la condensation de l'ADN. Dans l'immunoprécipitation de la chromatine couplée au séquençage de l'ADN ( ChIP-Seq ), HU ne révèle aucun événement de liaison spécifique. Au lieu de cela, il affiche une liaison uniforme à travers le génome reflétant vraisemblablement sa liaison principalement faible et non spécifique à la séquence, masquant ainsi la liaison de haute affinité in vivo .
Dans les souches dépourvues d'HU, le nucléoïde est "décondensé", ce qui correspond à un rôle de l'HU dans la compaction de l'ADN. Les études in vitro suivantes suggèrent des mécanismes possibles de la façon dont l'HU pourrait condenser et organiser l'ADN in vivo . Non seulement HU se lie de manière stable à l'ADN déformé avec des courbures, il induit des courbures flexibles même dans un ADN linéaire à une concentration inférieure à 100 nM. En revanche, HU montre l'effet architectural opposé sur l'ADN à des concentrations physiologiquement pertinentes plus élevées. Il forme des filaments de nucléoprotéines rigides provoquant le rétrécissement de l'ADN et non la flexion. Les filaments peuvent en outre former un réseau d'ADN (groupement d'ADN) extensible à la fois latéralement et médialement en raison de la multimérisation HU-HU déclenchée par la liaison à l'ADN non spécifique de la séquence.
Comment ces comportements d'HU sont-ils pertinents à l'intérieur de la cellule ? La formation de filaments nécessite une liaison à haute densité de HU sur l'ADN, un dimère de HU pour 9-20 pb d'ADN. Mais il n'y a qu'un dimère HU tous les ~150 pb de l'ADN chromosomique sur la base de l'abondance estimée de 30 000 dimères HU par cellule (4600000 pb / 30 000). Cela indique que les courbures flexibles sont plus susceptibles de se produire in vivo . La flexion flexible provoquerait une condensation due à une réduction de la longueur de persistance de l'ADN, comme le montrent les expériences de pinces magnétiques , qui permettent d'étudier la condensation d'une seule molécule d'ADN par une protéine de liaison à l'ADN. Cependant, en raison de la coopérativité , des filaments et des réseaux rigides pourraient se former dans certaines régions du chromosome. La formation de filaments à elle seule n'induit pas de condensation, mais la mise en réseau ou le regroupement d'ADN peut contribuer de manière substantielle à la condensation en rassemblant des segments de chromosomes distants ou proches.
IHF
Le facteur hôte d'intégration (IHF) est structurellement presque identique à HU mais se comporte différemment de HU à bien des égards. Contrairement à HU, qui se lie préférentiellement à un motif structurel quelle que soit la séquence, l'IHF se lie préférentiellement à une séquence d'ADN spécifique même si la spécificité résulte de la structure et de la déformabilité de l'ADN dépendantes de la séquence. La liaison spécifique de l'IHF aux sites apparentés plie fortement l'ADN de > 160 degrés. Une occurrence du motif de séquence apparentée est d'environ 3000 dans le génome d' E. coli . L'abondance estimée de l'IHF dans la phase de croissance est d'environ 6000 dimères par cellule. En supposant qu'un dimère IHF se lie à un seul motif et que le nucléoïde contienne plus d'un équivalent génomique pendant la phase de croissance exponentielle, la plupart des molécules IHF occuperaient des sites spécifiques dans le génome et ne condenseraient probablement l'ADN qu'en induisant une forte courbure.
Outre la liaison préférentielle à une séquence d'ADN spécifique, l'IHF se lie également à l'ADN d'une manière non spécifique à la séquence avec des affinités similaires à HU. Un rôle de la liaison non spécifique de l'IHF dans la condensation de l'ADN semble être critique dans la phase stationnaire, car l'abondance de l'IHF est multipliée par cinq dans la phase stationnaire et les dimères supplémentaires de l'IHF se lieraient probablement de manière non spécifique à l'ADN chromosomique. Contrairement à HU, IHF ne forme pas de filaments rigides épais à des concentrations plus élevées. Au lieu de cela, sa liaison non spécifique induit également une courbure de l'ADN bien que le degré de courbure soit beaucoup plus petit que celui de sites spécifiques et soit similaire à la courbure flexible induite par HU dans un ADN linéaire à de faibles concentrations. In vitro , la courbure induite par la liaison non spécifique de l'IHF peut provoquer une condensation de l'ADN et favorise la formation de complexes nucléoprotéiques d'ordre supérieur en fonction des concentrations de chlorure de potassium et de chlorure de magnésium. L'organisation de l'ADN d'ordre supérieur par l'IHF in vivo n'est pas encore claire.
H-NS
Une caractéristique distinctive de la protéine de structuration nucléoïde de type histone ou thermostable (H-NS) des autres NAP est la capacité de passer de la forme homodimérique à des concentrations relativement faibles (<1 x 10 -5 M) à un état oligomérique à des concentrations plus élevées. niveaux. En raison des propriétés d'oligomérisation, H-NS se propage latéralement le long de l'ADN riche en AT dans une réaction de nucléation , où les sites de haute affinité fonctionnent comme des centres de nucléation. La propagation de H-NS sur l'ADN entraîne deux résultats opposés en fonction de la concentration en magnésium dans la réaction. À faible concentration en magnésium (< 2 mM), H-NS forme des filaments de nucléoprotéines rigides alors qu'il forme des ponts inter- et intra-moléculaires à des concentrations plus élevées en magnésium (> 5 mM). La formation de filaments rigides entraîne un redressement de l'ADN sans condensation alors que le pontage provoque un repliement substantiel de l'ADN. L'analyse de la liaison de H-NS dans le génome par des tests ChIP-Seq a fourni une preuve indirecte de la propagation de H-NS sur l'ADN in vivo . H-NS se lie sélectivement à 458 régions du génome. Bien qu'il ait été démontré que H-NS préfère l'ADN incurvé formé par des pistes A répétées dans des séquences d'ADN, la base de la liaison sélective est la présence d'un motif de séquence conservé trouvé dans les régions riches en AT. Plus important encore, l'occurrence fréquente du motif de séquence dans une région de liaison H-NS qui peut renforcer les interactions protéine-protéine coopératives, et la longueur inhabituellement longue de la région de liaison sont compatibles avec la propagation de la protéine. Que la formation de filaments ou le pontage de l'ADN soit répandu in vivo dépend de la concentration physiologique de magnésium à l'intérieur de la cellule. Si la concentration en magnésium est uniformément faible (< 5 mM), H-NS formerait des filaments de nucléoprotéines rigides in vivo . Alternativement, s'il y a une répartition inégale du magnésium dans la cellule, cela pourrait favoriser à la fois le pontage et le raidissement de l'ADN, mais dans différentes régions du nucléoïde.
De plus, H-NS est surtout connu comme un silencieux génique global qui inhibe préférentiellement la transcription des gènes transférés horizontalement et c'est le filament rigide qui conduit au silençage génique. Dans l'ensemble, il semble que la formation de filaments rigides soit le résultat le plus probable des interactions H-NS-ADN in vivo qui conduisent à l'extinction des gènes mais n'induisent pas la condensation de l'ADN. Constamment, l'absence de H-NS ne modifie pas le volume nucléoïde. Cependant, il est possible qu'E. coli présente une concentration élevée de magnésium dans certaines conditions environnementales. Dans de telles conditions, H-NS peut passer de sa forme induisant un filament à la forme induisant un pont qui contribue à la condensation et à l'organisation de l'ADN.
Fis
Le facteur de stimulation par inversion (Fis) est une protéine de liaison à l'ADN spécifique à une séquence qui se lie à des séquences d'ADN spécifiques contenant un motif symétrique de 15 pb. Comme l'IHF, Fis induit une courbure de l'ADN sur des sites apparentés. La capacité de plier l'ADN est apparente dans la structure de l'homodimère Fis. Un homodimère Fis possède deux motifs hélice-tour-hélice (HTH), un pour chaque monomère. Un motif HTH reconnaît généralement le sillon majeur de l'ADN. Toutefois, la distance entre les hélices de reconnaissance d'ADN des deux HTH motifs dans l'homodimère Fis est de 25 Å , qui est ~ 8 Å plus courte que la hauteur d'une canonique B-ADN , ce qui indique que la protéine doit se plier ou tordre l' ADN de se lier de manière stable . De manière cohérente, la structure cristalline des complexes Fis-ADN montre que la distance entre les hélices de reconnaissance reste inchangée alors que l'ADN se courbe dans la plage de 60 à 75 degrés. Il existe 1464 régions de liaison Fis réparties dans le génome d' E. coli et un motif de liaison, identifié par ordinateur, correspond au motif connu de 15 pb. La liaison spécifique de Fis à de tels sites induirait des courbures dans l'ADN, contribuant ainsi à la condensation de l'ADN en réduisant la longueur de persistance de l'ADN. En outre, de nombreux sites de liaison Fis se produisent en tandem, tels que ceux des promoteurs d'ARN stables, par exemple le promoteur Pl de l' opéron d' ARNr rrnB . La courbure cohérente de Fis sur les sites en tandem est susceptible de créer une micro-boucle d'ADN qui peut contribuer davantage à la condensation de l'ADN.
Outre la liaison spécifique de haute affinité aux sites apparentés, Fis peut se lier à une séquence d'ADN aléatoire. La liaison à l'ADN non spécifique est importante car Fis est aussi abondant que HU dans la phase de croissance . Par conséquent, la plupart des molécules Fis devraient se lier à l'ADN d'une manière non spécifique à la séquence. Des expériences avec des pincettes magnétiques montrent que cette liaison non spécifique de Fis peut contribuer à la condensation et à l'organisation de l'ADN. Fis provoque une légère condensation d'une seule molécule d'ADN à < 1 mM, mais induit un repliement substantiel par la formation de boucles d'ADN d'une taille moyenne d'environ 800 pb à > 1 mM. Les boucles dans les expériences de pincettes magnétiques sont distinctes des micro-boucles créées par la courbure cohérente de l'ADN au niveau des sites apparentés, car elles nécessitent la formation de complexes ADN-protéine de haute densité obtenus par liaison indépendante de la séquence. Bien que l'apparition de telles boucles in vivo reste à démontrer, la liaison à haute densité de Fis peut se produire in vivo par l'action concertée de liaison à la fois spécifique et non spécifique. L'apparition en tandem de sites spécifiques pourrait initier une réaction de nucléation similaire à celle de H-NS, puis une liaison non spécifique conduirait à la formation de réseaux Fis localisés à haute densité. Le pontage entre ces régions localisées peut créer de grandes boucles d'ADN. Fis est présent exclusivement en phase de croissance et non en phase stationnaire . Ainsi, tout rôle dans la condensation chromosomique par Fis doit être spécifique aux cellules en croissance.
ARN associés aux nucléoïdes (naARN)
Les premières études examinant l'effet du traitement à la RNase A sur des nucléoïdes isolés ont indiqué que l' ARN participait à la stabilisation du nucléoïde à l'état condensé. De plus, le traitement avec la RNase A a rompu les fibres d'ADN en fibres plus fines, comme observé par une microscopie à force atomique du nucléoïde en utilisant la "procédure de lyse sur substrat". Ces découvertes ont démontré la participation de l'ARN dans la structure nucléoïde, mais l'identité de la ou des molécules d'ARN est restée inconnue jusqu'à récemment. La plupart des études sur HU se sont concentrées sur sa liaison à l'ADN. Cependant, HU se lie également aux hybrides ARNdb et ARN-ADN avec une affinité inférieure similaire à celle d'un ADNdb linéaire. De plus, HU se lie de préférence à des structures secondaires contenant de l'ARN et à un hybride ARN-ADN dans lequel l'ARN contient une entaille ou un surplomb. Les affinités de liaison de l'HU avec ces substrats d'ARN sont similaires à celles avec lesquelles elle se lie à l'ADN déformé. Une immunoprécipitation de l'ARN lié à l'HU couplée à une étude de transcription inverse et de microarray (RIP-Chip) ainsi qu'une analyse de l'ARN à partir de nucléoïdes intacts purifiés ont identifié des molécules d'ARN associées aux nucléoïdes qui interagissent avec l'HU. Plusieurs d'entre eux sont des ARN non codants, et l'un de ces ARN nommé naRNA4 (nucleoid-associated RNA 4) est codé dans un palindrome extragénique répétitif ( REP325 ). Dans une souche dépourvue de REP325 , le nucléoïde est décondensé comme il l'est dans une souche dépourvue de HU. naRNA4 participe très probablement à la condensation de l'ADN en connectant des segments d'ADN en présence de HU. Des études récentes fournissent des informations sur le mécanisme moléculaire de la façon dont naRNA4 établit des connexions ADN-ADN. L'ARN cible des régions d'ADN contenant des structures cruciformes et forme un complexe ARN-ADN essentiel à l'établissement de connexions ADN-ADN. Étonnamment, bien que l'HU aide à la formation du complexe, il n'est pas présent dans le complexe final, indiquant son rôle potentiel de catalyseur (chaperon). La nature du complexe ARN-ADN reste déroutante car la formation du complexe n'implique pas un appariement de bases Watson/Crick étendu mais est sensible à la RNase H, qui clive l'ARN dans un hybride ARN-ADN et le complexe se lie à un anticorps spécifique de Hybrides ARN-ADN.
Super-enroulement
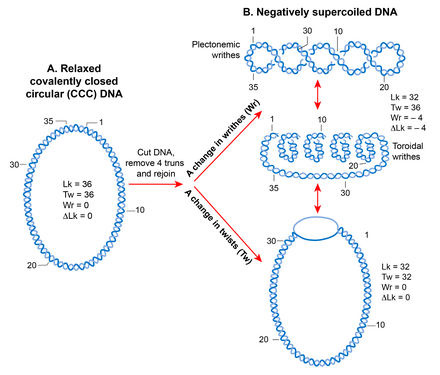
En raison de sa structure hélicoïdale , une molécule d'ADN double brin devient topologiquement contrainte sous la forme circulaire fermée de manière covalente, ce qui élimine la rotation des extrémités libres. Le nombre de fois où les deux brins se croisent dans un ADN topologiquement contraint est appelé nombre de liaison (Lk), qui équivaut au nombre de tours ou de torsions hélicoïdales dans une molécule circulaire. Le Lk d'un ADN topologique reste invariant, quelle que soit la façon dont la molécule d'ADN est déformée, tant qu'aucun des deux brins n'est rompu.
La Lk de l'ADN sous forme relâchée est définie comme Lk 0 . Pour tout ADN, Lk 0 peut être calculé en divisant la longueur (en pb) de l'ADN par le nombre de pb par tour d'hélice. Ceci est égal à 10,4 pb pour l' ADN de forme B relaxé . Tout écart par rapport à Lk 0 provoque un surenroulement dans l'ADN. Une diminution du nombre de liaison (Lk<Lk 0 ) crée un surenroulement négatif alors qu'une augmentation du nombre de liaison (Lk>Lk 0 ) crée un surenroulement positif.
L'état superenroulé (lorsque Lk n'est pas égal à Lk 0 ) entraîne une transition dans la structure de l'ADN qui peut se manifester par un changement du nombre de torsions (négatif < 10,4 pb/tour, positif > 10,4 pb par tour) et/ou en la formation de contorsions , appelées supercoils. Ainsi, Lk est défini mathématiquement comme une somme dépendante du signe des deux paramètres géométriques, la torsion et la torsion. Une mesure quantitative du superenroulement qui est indépendante de la taille des molécules d'ADN est la densité de superenroulement (σ) où σ =∆Lk/Lk 0 .
Writhes peut adopter deux structures; plectonème et solénoïde ou tore. Une structure plétonémique résulte de l'enchevêtrement de l'axe hélicoïdal. Les superbobines toroïdales naissent lorsque l'ADN forme plusieurs spirales, autour d'un axe et ne se croisant pas, comme celles d'un cordon téléphonique. Les contorsions sous la forme de plétonèmes sont respectivement droite et gauche dans un ADN superenroulé positivement ou négativement. La maniabilité des superbobines toroïdales est opposée à celles des pletonèmes. Les plétonèmes et les superbobines toroïdales peuvent être soit sous une forme libre, soit sous une forme liée avec des protéines. Le meilleur exemple du superenroulement toroïdal lié en biologie est le nucléosome eucaryote dans lequel l'ADN s'enroule autour des histones .
Superbobines plétonémiques dans E. coli

Dans la plupart des bactéries, l'ADN est présent sous forme superenroulée. La nature circulaire du chromosome d' E. coli en fait une molécule à contrainte topologique qui est principalement surenroulée négativement avec une densité moyenne estimée de superenroulement (σ) de -0,05. Dans la chromatine eucaryote , l'ADN se trouve principalement sous la forme toroïdale qui est restreinte et définie par des histones grâce à la formation de nucléosomes. En revanche, dans le nucléoïde d' E. coli , environ la moitié de l'ADN chromosomique est organisée sous la forme de superbobines libres et plectonémiques. L'ADN restant est retenu soit sous la forme plétonémique, soit sous des formes alternatives, y compris, mais sans s'y limiter, la forme toroïdale, par interaction avec des protéines telles que les NAP. Ainsi, les superenroulements plétonémiques représentent un superenroulement efficace du génome d' E. coli qui est responsable de sa condensation et de son organisation. Le superenroulement plétonémique et toroïdal aide à la condensation de l'ADN. Il est à noter qu'en raison de la ramification des structures plétonémiques, il fournit moins de condensation d'ADN que la structure toroïdale. Une molécule d'ADN de même taille avec des densités de superenroulement égales est plus compacte sous une forme toroïdale que sous une forme plétonémique. En plus de condenser l'ADN, le superenroulement aide à l'organisation de l'ADN. Il favorise le démêlage de l'ADN en réduisant la probabilité de caténation. Le superenroulement aide également à rapprocher deux sites distants d'ADN, favorisant ainsi une interaction fonctionnelle potentielle entre différents segments d'ADN.
Sources de superenroulement chez E. coli
Trois facteurs contribuent à générer et à maintenir le surenroulement de l'ADN chromosomique chez E. coli : (i) les activités des topoisomérases , (ii) l'acte de transcription et (iii) les NAP.
Topoisomérases
Les topoisomérases sont une catégorie particulière d'enzymes métaboliques de l'ADN qui créent ou suppriment le superenroulement en cassant puis en religuant des brins d'ADN. E. coli possède quatre topoisomérases. L'ADN gyrase introduit un superenroulement négatif en présence d'ATP et supprime le superenroulement positif en l'absence d'ATP. Dans toutes les formes de vie, l'ADN gyrase est la seule topoisomérase qui peut créer un superenroulement négatif et c'est en raison de cette capacité unique que les génomes bactériens possèdent des superenroulements négatifs libres ; L'ADN gyrase est présente dans toutes les bactéries mais absente des eucaryotes supérieurs. En revanche, Topo I s'oppose à l'ADN gyrase en relaxant l'ADN surenroulé négativement. Il existe des preuves génétiques suggérant qu'un équilibre entre les activités opposées de l'ADN gyrase et de Topo I est responsable du maintien d'un niveau stable de superhélicité négative moyenne dans E. coli . Les deux enzymes sont essentielles à la survie d' E. coli . Une souche nulle de topA , le gène codant pour Topo I, ne survit qu'en raison de la présence de mutations suppressives dans les gènes codant pour l'ADN gyrase. Ces mutations entraînent une réduction de l'activité de la gyrase, suggérant qu'un excès de surenroulement négatif dû à l'absence de Topo I est compensé par une réduction de l'activité de surenroulement négatif de l'ADN gyrase. Topo III est superflu dans E. coli et n'est pas connu pour avoir un rôle dans le surenroulement dans E. coli. La fonction principale de Topo IV est de résoudre les chromosomes frères. Cependant, il a été démontré qu'il contribuait également au niveau d'équilibre du superenroulement négatif en relaxant le superenroulement négatif avec Topo I.
| Topoisomérase | Taper | Fonction | Clivage simple ou double brin |
|---|---|---|---|
| Topoisomérase I | AI | Supprime (-) le superenroulement | SS |
| Topoisomérase III | AI | Supprime (-) le superenroulement | SS |
| Topoisomérase IV | IIA | Supprime (-) le superenroulement | DS |
| ADN-gyrase | IIA | Crée (-) supercoiling et supprime (+) supercoiling | DS |
Transcription
Un modèle de domaine de superenroulement jumeau proposé par Liu et Wang a fait valoir que le déroulement de la double hélice d' ADN pendant la transcription induit un superenroulement dans l'ADN, comme indiqué dans. Selon leur modèle, la transcription de l' ARN polymérase (RNAP) glissant le long de l'ADN force l'ADN à tourner sur son axe hélicoïdal . Un obstacle à la libre rotation de l'ADN pourrait survenir en raison d'une contrainte topologique, provoquant une surtorsion de l'ADN devant RNAP (positivement surenroulé) et l'ADN derrière RNAP devenant sous-torsadé (négativement surenroulé). Il a été constaté qu'une contrainte topologique n'est pas nécessaire car RNAP génère un couple suffisant qui provoque un superenroulement même dans une matrice d'ADN linéaire. Si l'ADN est déjà surenroulé négativement, cette action détend les superbobines négatives existantes avant de provoquer une accumulation de superbobines positives avant RNAP et introduit plus de superbobines négatives derrière RNAP. En principe, l'ADN gyrase et Topo I devraient éliminer respectivement les superenroulements positifs et négatifs en excès, mais si le taux d'élongation de l'ARNP dépasse le renouvellement des deux enzymes, la transcription contribue au niveau de superenroulement à l'état d'équilibre.
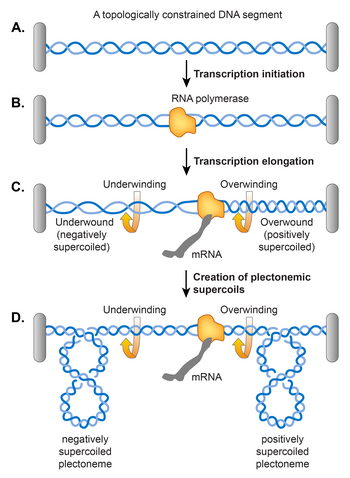
Contrôle du superenroulement par NAPs
Dans la chromatine eucaryote, l'ADN est rarement présent sous la forme libre superenroulée car les nucléosomes restreignent presque tous les superenroulements négatifs grâce à une liaison étroite de l'ADN aux histones. De même, dans E. coli , les complexes nucléoprotéiques formés par les NAP restreignent la moitié de la densité de superenroulement du nucléoïde. En d'autres termes, si une NAP se dissocie d'un complexe nucléoprotéique , l'ADN adopterait la forme libre et plétonémique. Il a été démontré expérimentalement que la liaison à l'ADN de HU, Fis et H-NS restreignait le surenroulement négatif dans un ADN détendu mais topologiquement contraint. Ils peuvent le faire soit en modifiant le pas hélicoïdal de l'ADN, soit en générant des contorsions toroïdales par pliage et enroulement de l'ADN. Alternativement, les NAP peuvent se lier de préférence à et stabiliser d'autres formes de l'ADN sous-enroulé telles que les structures cruciformes et les plétonèmes ramifiés. Il a été rapporté que Fis organise des plétonèmes ramifiés par sa liaison aux régions de croisement et que HU se lie préférentiellement aux structures cruciformes.
Les NAP régulent également indirectement le superenroulement de l'ADN. Fis peut moduler le superenroulement en réprimant la transcription des gènes codant l'ADN gyrase. Il existe des preuves génétiques suggérant que l'HU contrôle les niveaux de superenroulement en stimulant l'ADN gyrase et en réduisant l'activité de Topo I. À l'appui des études génétiques, il a été démontré que l'HU stimule la décaténation de l'ADN catalysée par l'ADN gyrase in vitro . On ne sait pas mécaniquement comment HU module les activités de la gyrase et de Topo I. HU pourrait interagir physiquement avec l'ADN gyrase et Topo I ou les activités d'organisation de l'ADN de HU telles que la courbure de l'ADN peuvent faciliter ou inhiber l'action de l'ADN gyrase et Topo I respectivement.
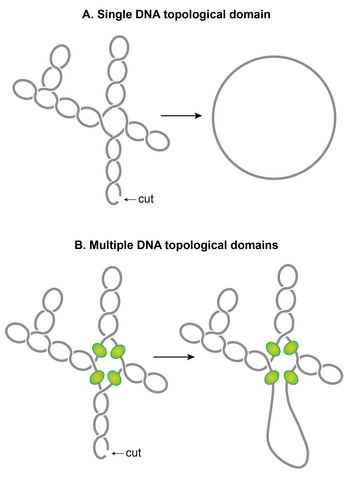
Les superbobines plétonémiques s'organisent en plusieurs domaines topologiques
L'une des caractéristiques frappantes du nucléoïde est que les superbobines plétonémiques sont organisées en plusieurs domaines topologiques. En d'autres termes, une seule coupe dans un domaine ne fera que détendre ce domaine et pas les autres. Un domaine topologique se forme à cause d'une barrière de superenroulement-diffusion. Des études indépendantes utilisant différentes méthodes ont rapporté que les domaines topologiques sont de taille variable allant de 10 à 400 ko. Un placement aléatoire de barrières couramment observé dans ces études semble expliquer la grande variabilité de la taille des domaines.
Bien que les identités des barrières de domaine restent à établir, les mécanismes possibles responsables de la formation des barrières incluent : (i) Une barrière de domaine pourrait se former lorsqu'une protéine ayant la capacité de retenir les supercoils se lie simultanément à deux sites distincts sur le chromosome formant une topologie boucle ou domaine d'ADN isolé. Il a été démontré expérimentalement que le bouclage à médiation protéique dans l'ADN superenroulé peut créer un domaine topologique. Les NAP tels que H-NS et Fis sont des candidats potentiels, en fonction de leurs capacités de bouclage de l'ADN et de la distribution de leurs sites de liaison. (ii) Les éléments de mosaïque intercalés bactériens (BIME) apparaissent également comme des candidats potentiels pour les barrières de domaine. Les BIME sont des séquences répétées palindromiques qui se trouvent généralement entre les gènes. Il a été démontré qu'un BIME empêche la diffusion du superenroulement dans une cassette topologique de conception synthétique insérée dans le chromosome d' E. coli . Il existe environ 600 BIME répartis dans le génome, divisant éventuellement le chromosome en 600 domaines topologiques. (iii) Les barrières pourraient également résulter de la fixation de l'ADN à la membrane cellulaire par une protéine qui se lie à la fois à l'ADN et à la membrane ou par la transcription naissante et la traduction de protéines ancrées dans la membrane. (iv) L'activité de transcription peut générer des barrières de superenroulement-diffusion. Il a été démontré qu'un RNAP à transcription active bloque la dissipation des superbobines plectonémiques, formant ainsi une barrière de diffusion de supercoiling.
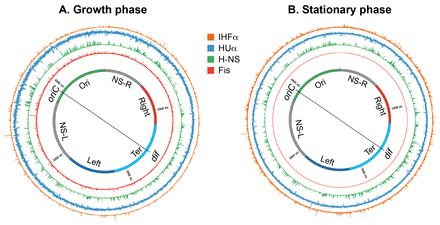
Dynamique nucléoïde dépendante de la phase de croissance
Le nucléoïde se réorganise en cellules en phase stationnaire, suggérant que la structure nucléoïde est hautement dynamique, déterminée par l'état physiologique des cellules. Une comparaison des cartes de contact à haute résolution du nucléoïde a révélé que les contacts à longue distance dans le macrodomaine Ter augmentaient dans la phase stationnaire , par rapport à la phase de croissance . De plus, les limites CID dans la phase stationnaire étaient différentes de celles trouvées dans la phase de croissance. Enfin, la morphologie nucléoïde subit une transformation massive au cours d'une phase stationnaire prolongée ; le nucléoïde présente des structures toroïdales ordonnées.
Des modifications spécifiques à la phase de croissance de la structure nucléoïde pourraient être provoquées par une modification des niveaux de protéines architecturales de l'ADN associées aux nucléoïdes (les sous-unités NAP et Muk), du superenroulement et de l'activité de transcription. L'abondance des NAP et des sous-unités Muk change en fonction du cycle de croissance bactérienne. Fis et la protéine de liaison à l'ADN induite par la famine Dps, une autre NAP, sont presque exclusivement présentes dans la phase de croissance et la phase stationnaire respectivement. Les niveaux de Fis augmentent lors de l'entrée en phase exponentielle, puis diminuent rapidement alors que les cellules sont encore en phase exponentielle, atteignant des niveaux indétectables en phase stationnaire. Alors que les niveaux de Fis commencent à baisser, les niveaux de Dps commencent à augmenter et atteignent un maximum en phase stationnaire. Une transition dramatique dans la structure nucléoïde observée dans la phase stationnaire prolongée a été principalement attribuée à Dps. Il forme des assemblages ADN/ cristallins qui agissent pour protéger le nucléoïde des agents endommageant l'ADN présents pendant la famine.
HU, IHF et H-NS sont présents à la fois en phase de croissance et en phase stationnaire. Cependant, leur abondance change de manière significative, de sorte que HU et Fis sont les NAP les plus abondants en phase de croissance, tandis que IHF et Dps deviennent les NAP les plus abondants en phase stationnaire. HUαα est la forme prédominante en phase exponentielle précoce, alors que la forme hétérodimère prédomine en phase stationnaire, avec des quantités mineures d'homodimères. Cette transition a des conséquences fonctionnelles sur la structure nucléoïde, car les deux formes semblent organiser et condenser l'ADN différemment ; les homo- et hétérodimères forment des filaments, mais seul l'homodimère peut rassembler plusieurs segments d'ADN pour former un réseau d'ADN. Le nombre de copies de MukB est multiplié par deux en phase stationnaire. Une augmentation du nombre de molécules MukB pourrait avoir une influence sur la processivité du complexe MukBEF en tant que facteur d'extrusion de boucles d'ADN, entraînant un nombre plus grand ou plus grand de boucles.
Le superenroulement peut agir de manière concertée avec les protéines architecturales de l'ADN pour réorganiser le nucléoïde. Le niveau global de superenroulement diminue dans la phase stationnaire, et le superenroulement présente un schéma différent au niveau régional. Les changements de superenroulement peuvent modifier l'organisation topologique du nucléoïde. De plus, étant donné qu'une région chromosomique d'activité de transcription élevée forme une frontière CID, des changements dans l'activité de transcription au cours des différentes phases de croissance pourraient modifier la formation de frontières CID, et donc l'organisation spatiale du nucléoïde. Il est possible que les changements dans les limites CID observés dans la phase stationnaire soient dus à l'expression élevée d'un ensemble différent de gènes dans la phase stationnaire par rapport à la phase de croissance.
Structure nucléoïde et expression des gènes
NAP et expression des gènes
La structure chromosomique d' E. coli et l'expression des gènes semblent s'influencer réciproquement. D'une part, une corrélation d'une limite CID avec une activité de transcription élevée indique que l'organisation des chromosomes est entraînée par la transcription. D'autre part, la structure 3D de l'ADN dans le nucléoïde à toutes les échelles peut être liée à l'expression des gènes. Premièrement, il a été montré que la réorganisation de l'architecture 3D du nucléoïde dans E. coli peut moduler dynamiquement le modèle de transcription cellulaire. Un mutant de HUa a rendu le nucléoïde très condensé par l'augmentation de la superhélicité positive de l'ADN chromosomique. Par conséquent, de nombreux gènes ont été réprimés et de nombreux gènes quiescents ont été exprimés. En outre, il existe de nombreux cas spécifiques dans lesquels des changements architecturaux locaux à médiation protéique altèrent la transcription des gènes. Par exemple, la formation de filaments nucléoprotéiques rigides par H-NS bloque l'accès de l'ARNP au promoteur et empêche ainsi la transcription du gène. Grâce au silençage génique, H-NS agit comme un répresseur global inhibant préférentiellement la transcription des gènes transférés horizontalement. Dans un autre exemple, la liaison spécifique de HU à l' opéron gal facilite la formation d'une boucle d'ADN qui maintient l' opéron gal réprimé en l'absence de l'inducteur. La micro-boucle d'ADN topologiquement distincte créée par la courbure cohérente de l'ADN par Fis au niveau des promoteurs d'ARN stables active la transcription. La courbure de l'ADN par l'IHF contrôle de manière différentielle la transcription à partir des deux promoteurs en tandem de l' opéron ilvGMEDA dans E. coli . Les changements topologiques spécifiques par les NAP régulent non seulement la transcription des gènes, mais sont également impliqués dans d'autres processus tels que l'initiation de la réplication de l'ADN, la recombinaison et la transposition. Contrairement à la régulation spécifique des gènes, il reste à déterminer comment la structure chromosomique d'ordre supérieur et sa dynamique influencent l'expression des gènes à l'échelle mondiale au niveau moléculaire.
Superenroulement de l'ADN et expression des gènes
Il existe une interconnexion bidirectionnelle entre le surenroulement de l'ADN et la transcription des gènes. Le surenroulement négatif de la région du promoteur peut stimuler la transcription en facilitant la fusion du promoteur et en augmentant l'affinité de liaison à l'ADN d'un régulateur protéique. Les éclats de transcription stochastiques semblent être une caractéristique générale des gènes fortement exprimés, et les niveaux de superenroulement de la matrice d'ADN contribuent à l'éclatement de la transcription. Selon le modèle de domaine de superenroulement jumeau, la transcription d'un gène peut influencer la transcription d'autres gènes voisins via un relais de superenroulement. Un tel exemple est l'activation du promoteur leu-500 . Le superenroulement n'intervient pas seulement dans les changements spécifiques aux gènes, mais il médie également les changements à grande échelle dans l'expression des gènes. L'organisation topologique du nucléoïde pourrait permettre l'expression indépendante de gènes sensibles au superenroulement dans différents domaines topologiques. Une carte à l'échelle du génome du superenroulement non restreint a montré que les régions génomiques ont différentes densités de superenroulement à l'état d'équilibre, indiquant que le niveau de superenroulement diffère dans les domaines topologiques individuels. En conséquence, un changement de superenroulement peut entraîner une expression génique spécifique à un domaine, en fonction du niveau de superenroulement dans chaque domaine.
L'effet du superenroulement sur l'expression des gènes peut être médié par les NAP qui influencent directement ou indirectement le superenroulement. L'effet de l'HU sur l'expression des gènes semble impliquer un changement dans le superenroulement et peut-être une organisation de l'ADN d'ordre supérieur. Une corrélation positive entre la liaison à l'ADN gyrase et la régulation à la hausse des gènes causée par l'absence de HU suggère que des changements dans le superenroulement sont responsables de l'expression différentielle. HU s'est également avéré responsable d'un effet positionnel sur l'expression des gènes en isolant les unités transcriptionnelles en limitant le superenroulement induit par la transcription. Des mutations ponctuelles dans HUa ont considérablement modifié le profil d'expression génique d' E. coli, modifiant sa morphologie , sa physiologie et son métabolisme . En conséquence, la souche mutante était plus invasive pour les cellules de mammifères. Cet effet dramatique était concomitant avec le compactage des nucléoïdes et l'augmentation du superenroulement positif. La protéine mutante était un octamère, contrairement au dimère de type sauvage. Il enveloppe l'ADN sur sa surface d'une manière droitière, retenant les superbobines positives par opposition à l'HU de type sauvage. Ces études montrent que les substitutions d'acides aminés dans HU peuvent avoir un effet dramatique sur la structure nucléoïde, qui à son tour entraîne des changements phénotypiques importants.
Étant donné que MukB et HU sont devenus des acteurs essentiels dans les interactions ADN à longue distance, il sera intéressant de comparer l'effet de chacune de ces deux protéines sur l'expression globale des gènes. Bien que HU semble contrôler l'expression des gènes en modulant la densité de superenroulement, le mécanisme moléculaire exact reste inconnu et l'impact de MukB sur l'expression des gènes doit encore être analysé.
Organisation spatiale
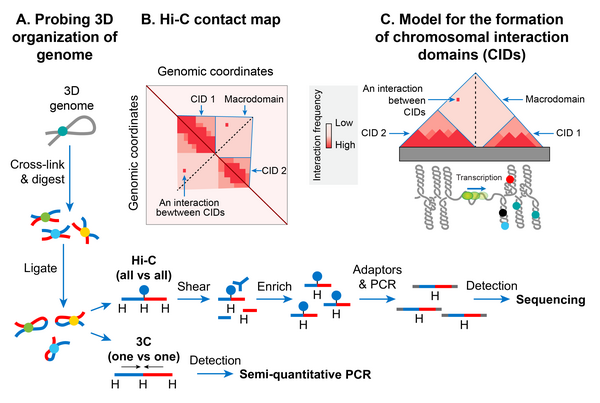
Domaines d'interaction chromosomique
Ces dernières années, l'avènement d'une méthode moléculaire appelée capture de conformation chromosomique (3C) a permis d'étudier une organisation spatiale à haute résolution des chromosomes chez les bactéries et les eucaryotes. 3C et sa version couplée au séquençage profond (Hi-C) déterminent la proximité physique, le cas échéant, entre deux loci génomiques quelconques dans l'espace 3D. Une carte de contact à haute résolution des chromosomes bactériens, y compris le chromosome d' E. coli, a révélé qu'un chromosome bactérien est segmenté en de nombreuses régions hautement interactives appelées domaines d'interaction chromosomique (CID). Les CID sont équivalents aux domaines d'association topologique (TAD) observés dans de nombreux chromosomes eucaryotes, suggérant que la formation de CID est un phénomène général d'organisation du génome. Deux caractéristiques définissent les CID ou les TAD. Premièrement, les régions génomiques d'un CID interagissent physiquement les unes avec les autres plus fréquemment qu'avec les régions génomiques en dehors de ce CID ou avec celles d'un CID voisin. Deuxièmement, la présence d'une frontière entre les CID qui empêche les interactions physiques entre les régions génomiques de deux CID voisins.
Le chromosome d' E. coli s'est avéré être constitué de 31 CID en phase de croissance. La taille des CID variait de 40 à ~ 300 ko. Il semble qu'une barrière de diffusion de superenroulement responsable de la ségrégation des boucles d'ADN plétonémiques en domaines topologiques fonctionne comme une frontière CID chez E. coli et de nombreuses autres bactéries. En d'autres termes, la présence d'une barrière de superenroulement-diffusion définit la formation de CID. Les découvertes du sondage Hi-C des chromosomes dans E. coli , Caulobacter crescentus et Bacillus subtilis convergent vers un modèle que les CID forment parce que la boucle plétonémique ainsi que les activités d'organisation de l'ADN des NAP favorisent les interactions physiques entre les loci génomiques, et une limite CID se compose de une région sans plectonème (PFR) qui empêche ces interactions. Un PFR est créé en raison d'une activité de transcription élevée, car le déroulement hélicoïdal de l'ADN en transcrivant activement l'ARNP retient les superbobines plétonémiques. En conséquence, la dissipation des superenroulements est également bloquée, créant une barrière de diffusion de superenroulement. La preuve indirecte de ce modèle provient d'une observation selon laquelle les CID de chromosomes bactériens, y compris le chromosome d' E. coli, présentent des gènes hautement transcrits à leurs frontières, indiquant un rôle de la transcription dans la formation d'une frontière CID. Des preuves plus directes sont venues de la découverte que le placement d'un gène hautement transcrit à une position où aucune limite n'était présente a créé une nouvelle limite CID dans le chromosome de C. crescentus . Cependant, toutes les limites CID ne sont pas corrélées avec des gènes hautement transcrits dans le chromosome d' E. coli, ce qui suggère que d'autres facteurs inconnus sont également responsables de la formation de limites CID et de barrières de diffusion super-enroulées.
Macrodomaines
Les boucles d'ADN plétonémiques organisées en domaines topologiques ou CID semblent fusionner davantage pour former de grands domaines spatialement distincts appelés macrodomaines (MD). Chez E. coli, les MD ont été initialement identifiés comme de grands segments du génome dont les marqueurs d'ADN se sont localisés ensemble (co-localisés) dans des études d' hybridation in situ en fluorescence (FISH). Une grande région génomique (~1-Mb) couvrant le locus oriC (origine de la réplication chromosomique) co-localisée et a été appelée macrodomaine Ori. De même, une grande région génomique (~1-Mb) couvrant la région terminale de réplication ( ter ) co-localisée et a été appelée macrodomaine Ter. Les MD ont ensuite été identifiés en fonction de la fréquence à laquelle les paires de sites lambda att insérés à divers endroits distants du chromosome se sont recombinées les unes avec les autres. Dans cette méthode basée sur la recombinaison, une DM a été définie comme une grande région génomique dont les sites d'ADN peuvent principalement se recombiner les uns avec les autres, mais pas avec ceux en dehors de cette DM. La méthode basée sur la recombinaison a confirmé les DM Ori et Ter qui ont été identifiés dans les études FISH et a identifié deux DM supplémentaires.
Les deux MD supplémentaires ont été formées par les régions supplémentaires de ~ 1 Mb flanquant le Ter et ont été appelées Gauche et Droite. Ces quatre MD (Ori, Ter, Left et Right) composaient la majeure partie du génome, à l'exception de deux régions génomiques flanquant l'Ori. Ces deux régions (NS-L et NS-R) étaient plus flexibles et non structurées par rapport à une MD car les sites d'ADN y étaient recombinés avec des sites d'ADN situés dans les MD des deux côtés. La position génétique d' oriC semble dicter la formation des DM, car le repositionnement d' oriC par manipulation génétique entraîne la réorganisation des DM. Par exemple, les régions génomiques les plus proches de l' oriC se comportent toujours comme un NS quelle que soit la séquence d'ADN et les régions plus éloignées se comportent toujours comme des MD.
La technique Hi-C a en outre confirmé une organisation spatiale hiérarchique des CID sous forme de macrodomaines. En d'autres termes, les CID d'un macrodomaine interagissaient physiquement les uns avec les autres plus fréquemment qu'avec les CID d'un macrodomaine voisin ou avec des loci génomiques en dehors de ce macrodomaine. Les données Hi-C ont montré que le chromosome d' E. coli se partageait en deux domaines distincts. La région entourant ter formait un domaine isolé qui chevauchait le Ter MD précédemment identifié. Les contacts ADN-ADN dans ce domaine n'ont eu lieu que dans la gamme allant jusqu'à ~ 280 kb. Le reste du chromosome formait un domaine unique dont les loci génomiques présentaient des contacts de l'ordre de > 280 kb. Alors que la plupart des contacts dans ce domaine étaient limités à une distance maximale de ~ 500 kb, il y avait deux régions lâches dont les loci génomiques formaient des contacts à des distances encore plus grandes (jusqu'à ~ 1 Mb). Ces régions lâches correspondaient aux régions flexibles et moins structurées (NS) précédemment identifiées. Les limites du domaine isolé englobant ter et les deux régions lâches identifiées par la méthode Hi-C ont segmenté le chromosome entier en six régions qui correspondent aux quatre régions MD et deux régions NS définies par des tests basés sur la recombinaison.
Protéines qui entraînent la formation de macrodomaines
MatP
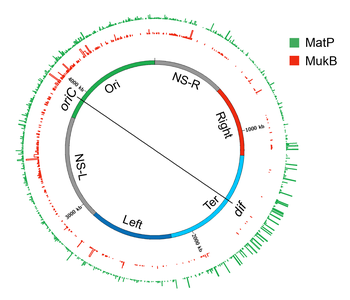
Une recherche de protéine(s) responsable(s) de la formation de macrodomaines a conduit à l'identification de la protéine Macrodomain Ter (MatP). MatP se lie presque exclusivement dans le Ter MD en reconnaissant un motif de 13 pb appelé séquence ter de macrodomaine ( matS ). Il y a 23 sites matS présents dans le domaine Ter, en moyenne il y a un site tous les 35 ko. Une preuve supplémentaire de la liaison de MatP dans le domaine Ter provient de l'imagerie par fluorescence de MatP. Des foyers MatP discrets ont été observés qui co-localisés avec des marqueurs d'ADN du domaine Ter. Un fort enrichissement du signal ChIP-Seq dans le Ter MD corrobore également la liaison préférentielle de MatP à ce domaine.
MatP condense l'ADN dans le domaine Ter car l'absence de MatP augmente la distance entre deux marqueurs d'ADN fluorescents situés à 100 kb l'un de l'autre dans le domaine Ter. De plus, MatP est un acteur essentiel pour isoler le domaine Ter du reste du chromosome. Il favorise les contacts ADN-ADN au sein du domaine Ter mais empêche les contacts entre les loci d'ADN du domaine Ter et ceux des régions flanquantes. Comment MatP condense-t-il l'ADN et favorise-t-il les contacts ADN-ADN ? Les résultats expérimentaux sont contradictoires. MatP peut former une boucle d'ADN entre deux sites matS in vitro et son activité de boucle d'ADN dépend de la tétramérisation de MatP. La tétramérisation se produit via des interactions en spirale entre deux molécules MatP liées à l'ADN. Un modèle évident basé sur des résultats in vitro est que MatP favorise les contacts ADN-ADN in vivo en pontant les sites matS . Cependant, bien que MatP ait connecté des sites distants dans les études Hi-C, il n'a pas spécifiquement connecté les sites matS . De plus, un mutant MatP incapable de former des tétramères s'est comporté comme du type sauvage. Ces résultats vont à l'encontre du modèle de pontage matS pour l'organisation Ter, laissant le mécanisme d'action MatP insaisissable. Une possibilité est que MatP se propage aux segments d'ADN voisins à partir de son site de liaison primaire matS et ponte des sites distants via un mécanisme qui ne dépend pas de la tétramérisation.
MukBEF
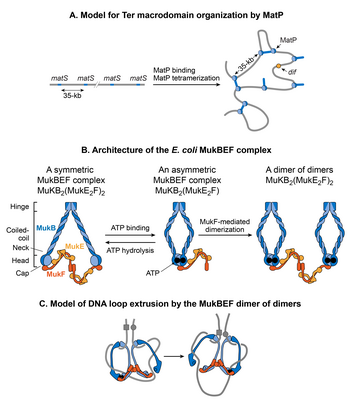
MukB appartient à une famille d'ATPases appelées maintenance structurelle des protéines chromosomiques (SMC), qui participent à l'organisation chromosomique d'ordre supérieur chez les eucaryotes. Deux monomères MukB s'associent via une interaction continue antiparallèle en spirale formant une tige rigide de 100 nm de long. Une région de charnière flexible se produit au milieu de la tige. En raison de la flexibilité de la région de charnière, MukB adopte une forme en V caractéristique de la famille SMC. Les sous-unités non SMC associées à MukB sont MukE et MukF. L'association ferme la formation en V, ce qui donne de grandes structures en forme d'anneau. MukE et MukF sont codés avec MukB dans le même opéron dans E. coli . La suppression de l'une ou l'autre des sous-unités donne le même phénotype, ce qui suggère que le complexe MukBEF est l'unité fonctionnelle in vivo . Les activités de liaison à l'ADN du complexe résident dans la sous-unité MukB, tandis que MukE et MukF modulent l'activité MukB.
Le complexe MukBEF, avec Topo IV, est requis pour la décaténation et le repositionnement des oriC nouvellement répliqués . Le rôle de MukBEF n'est pas limité lors de la réplication de l'ADN. Il organise et condense l'ADN même dans les cellules qui ne se répliquent pas. La récente carte de conformation chromosomique à haute résolution de la souche E. coli appauvrie en MukB révèle que MukB participe à la formation d'interactions ADN-ADN sur l'ensemble du chromosome, sauf dans le domaine Ter. Comment MukB est-il empêché d'agir dans le domaine Ter ? MatP interagit physiquement avec MukB, empêchant ainsi MukB de se localiser dans le domaine Ter. Ceci est évident dans la liaison à l'ADN de MatP et MukB dans le domaine Ter. La liaison à l'ADN de MatP est enrichie dans le domaine Ter, tandis que la liaison à l'ADN de MukB est réduite par rapport au reste du génome. De plus, dans une souche déjà dépourvue de MatP, l'absence de MukB entraîne une réduction des contacts ADN dans tout le chromosome, y compris le domaine Ter. Ce résultat est en accord avec le point de vue selon lequel MatP déplace MukB du domaine Ter.
Comment fonctionne le complexe MukBEF pour organiser le chromosome d' E. coli ? Selon l'opinion actuelle, les complexes SMC organisent les chromosomes en extrudant des boucles d'ADN. Les complexes SMC effectuent une translocation le long de l'ADN pour extruder des boucles de manière cis (sur la même molécule d'ADN), dans laquelle la taille des boucles dépend de la processivité du complexe. Les complexes SMC de différents organismes diffèrent par le mécanisme d'extrusion de la boucle. La microscopie à fluorescence d'une seule molécule de MukBEF dans E. coli suggère que l'unité fonctionnelle minimale in vivo est un dimère de dimères. Cette unité est formée par la jonction de deux complexes MukBEF liés à l'ATP par dimérisation médiée par MukF. MukBEF se localise dans la cellule sous forme de 1 à 3 grappes allongées parallèlement au grand axe de la cellule. Chaque cluster contient en moyenne ~ 8-10 dimères de dimères. Selon le modèle actuel, le MukBEF extrude des boucles d'ADN de manière « escalade ». Un dimère des dimères libère un segment d'ADN et capture un nouveau segment d'ADN sans se dissocier du chromosome. Outre le bouclage de l'ADN, un lien entre le superenroulement négatif et la fonction MukBEF in vivo ainsi que la capacité de la sous-unité MukB à contraindre les superenroulements négatifs in vitro suggère que MukBEF organise l'ADN en générant des superenroulements.
Rôle des NAP et des naRNA
En plus de contribuer à la compaction des chromosomes en pliant, en pontant et en bouclant l'ADN à une plus petite échelle (~ 1 kb), les NAP participent à la condensation et à l'organisation de l'ADN en favorisant les contacts ADN-ADN à longue distance. Deux NAP, Fis et HU, ont émergé comme les acteurs clés dans la promotion des contacts ADN-ADN à longue distance qui se produisent tout au long du chromosome. Il reste à étudier comment les activités d'organisation de l'ADN de Fis et HU qui sont bien comprises à une plus petite échelle (~ 1 kb) entraînent la formation d'interactions ADN-ADN à longue portée. Néanmoins, certaines des interactions d'ADN médiées par HU nécessitent la présence de naRNA4. naRNA4 participe également à l'établissement de contacts ADN à longue distance. HU catalyse certains des contacts, pas tous, ce qui suggère que l'ARN participe avec d'autres NAP à la formation de contacts avec l'ADN. HU semble également agir avec MukB pour favoriser les interactions ADN-ADN à longue distance. Ce point de vue est basé sur des observations selon lesquelles l'absence de HU ou de MukB a entraîné une réduction des mêmes contacts ADN-ADN. On ne sait pas comment MukB et HU agissent ensemble pour favoriser les interactions ADN-ADN. Il est possible que les deux protéines interagissent physiquement. Alternativement, alors que MukBEF extrude de grandes boucles d'ADN, HU condense et organise ces boucles.
Il existe des rapports selon lesquels les gènes d' E. coli liés fonctionnellement sont physiquement ensemble dans l'espace 3-D à l'intérieur du chromosome même s'ils sont éloignés les uns des autres par la distance génétique. La proximité spatiale des gènes fonctionnellement liés non seulement rendrait les fonctions biologiques plus compartimentées et efficaces, mais contribuerait également au repliement et à l'organisation spatiale du nucléoïde. Une étude récente utilisant des marqueurs fluorescents pour la détection de loci d'ADN spécifiques a examiné les distances physiques par paires entre les sept opérons d'ARNr qui sont génétiquement séparés les uns des autres (jusqu'à deux millions de pb). Il a signalé que tous les opérons, à l'exception de rrn C, étaient à proximité physique. Étonnamment, les études 3C-seq n'ont pas révélé le regroupement physique des opérons rrn , ce qui contredit les résultats de l'étude basée sur la fluorescence. Par conséquent, une enquête plus approfondie est nécessaire pour résoudre ces observations contradictoires. Dans un autre exemple, GalR forme un réseau d'interaction de sites de liaison GalR qui sont dispersés à travers le chromosome. GalR est un régulateur transcriptionnel du régulon du galactose composé de gènes codant pour des enzymes pour le transport et le métabolisme du sucre D-galactose. GalR n'existe que dans un ou deux foyers dans les cellules et peut s'auto-assembler en de grandes structures ordonnées. Par conséquent, il semble que GalR lié à l'ADN se multimérise pour former des interactions à longue distance.
Forme et structure globales
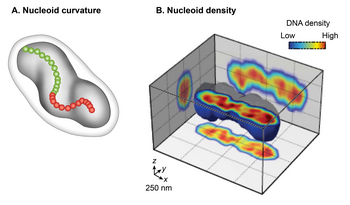
La microscopie électronique à transmission conventionnelle (MET) de cellules E. coli fixées chimiquement a représenté le nucléoïde comme un organite de forme irrégulière . Cependant, l' imagerie par fluorescence à grand champ de nucléoïdes vivants en 3D a révélé une forme ellipsoïde discrète. La superposition d'une image en contraste de phase de la cellule et de l'image fluorescente du nucléoïde a montré une juxtaposition étroite uniquement dans la dimension radiale sur toute sa longueur du nucléoïde à la périphérie de la cellule. Cette découverte indique un confinement radial du nucléoïde. Un examen détaillé de l'image de fluorescence 3D après coupe transversale perpendiculaire à son axe long a en outre révélé deux caractéristiques globales du nucléoïde : la courbure et les régions longitudinales à haute densité. L'examen de la chiralité de la ligne médiane du nucléoïde en connectant le centre d'intensité de chaque section transversale a montré que la forme globale du nucléoïde est incurvée. La distribution de l'intensité de fluorescence dans les sections transversales a révélé une sous-structure de densité, constituée de régions ou de faisceaux courbes à haute densité au cœur central et de régions à faible densité à la périphérie. Une implication du confinement radial est qu'il détermine la forme courbe du nucléoïde. Selon un modèle, le nucléoïde est contraint de se plier car il est confiné dans une cellule cylindrique d' E. coli dont le rayon est inférieur à sa longueur pliable (longueur de persistance). Ce modèle était soutenu par des observations selon lesquelles l'élimination de la paroi cellulaire ou l'inhibition de la synthèse de la paroi cellulaire augmentait le rayon de la cellule et entraînait une augmentation concomitante du rayon hélicoïdal et une diminution du pas hélicoïdal dans le nucléoïde.
Connexions nucléoïde-membrane
Une force d'expansion due aux connexions ADN-membrane semble fonctionner en opposition aux forces de condensation pour maintenir un niveau de condensation optimal du nucléoïde. Les études de fractionnement cellulaire et de microscopie électronique ont d'abord indiqué la possibilité de connexions ADN-membrane. Il existe maintenant plusieurs exemples connus de connexions ADN-membrane. La transition est un mécanisme de transcription, de traduction et d'insertion simultanées de protéines membranaires naissantes qui forment des contacts ADN-membrane transitoires. Il a été démontré que la transition de deux protéines membranaires LacY et TetA provoque le repositionnement des loci chromosomiques vers la membrane. Un autre mécanisme des connexions nucléoïde-membrane est le contact direct entre les régulateurs de transcription ancrés dans la membrane et leurs sites cibles dans le chromosome. Un exemple de régulateur de transcription dans E. coli est CadC. CadC contient un domaine sensoriel périplasmique et un domaine de liaison à l'ADN cytoplasmique. La détection d'un environnement acide par son domaine sensoriel périplasmique stimule l'activité de liaison à l'ADN de CadC, qui active alors la transcription de ses gènes cibles. La localisation membranaire des gènes régulés par un régulateur de transcription membranaire reste à démontrer. Néanmoins, l'activation de gènes cibles dans le chromosome par ces régulateurs devrait entraîner un contact nucléoïde-membrane bien qu'il s'agisse d'un contact dynamique. Outre ces exemples, le chromosome est également spécifiquement ancré à la membrane cellulaire par une interaction protéine-protéine entre des protéines liées à l'ADN, par exemple SlmA et MatP, et le divisome . Étant donné que les gènes codant pour les protéines membranaires sont distribués dans tout le génome, les contacts dynamiques ADN-membrane par transfert peuvent agir comme une force d'expansion nucléoïde. Cette force d'expansion fonctionnerait en opposition aux forces de condensation pour maintenir un niveau de condensation optimal. La formation de nucléoïdes hautement condensés lors de l'exposition des cellules d' E. coli au chloramphénicol, qui bloque la traduction, soutient la force d'expansion des contacts ADN-membrane transitoires formés par la transition. La forme ronde des nucléoïdes trop condensés après traitement au chloramphénicol suggère également un rôle pour les contacts ADN-membrane médiés par la transition dans la définition de la forme ellipsoïde du nucléoïde.
Visualisation
Le nucléoïde peut être clairement visualisé sur une micrographie électronique à très fort grossissement , où, bien que son apparence puisse différer, il est clairement visible contre le cytosol . Parfois, même des brins de ce que l'on pense être de l' ADN sont visibles. En colorant avec le colorant Feulgen , qui colore spécifiquement l'ADN, le nucléoïde peut également être vu au microscope optique . L' ADN intercalant taches DAPI et le bromure d' éthidium sont largement utilisés pour la microscopie par fluorescence de nucléoïdes. Il a une forme irrégulière et se trouve dans les cellules procaryotes.
Dommages à l'ADN et réparation
Des changements dans la structure du nucléoïde des bactéries et des archées sont observés après exposition à des conditions endommageant l'ADN. Les nucléoïdes des bactéries Bacillus subtilis et Escherichia coli deviennent tous deux significativement plus compacts après irradiation UV. La formation de la structure compacte dans E. coli nécessite l' activation de RecA via des interactions RecA-ADN spécifiques. La protéine RecA joue un rôle clé dans la réparation par recombinaison homologue des dommages à l'ADN.
Comme pour B. subtilis et E. coli ci-dessus, les expositions de l' archéon Haloferax volcanii à des stress qui endommagent l'ADN provoquent un compactage et une réorganisation du nucléoïde. La compaction dépend du complexe protéique Mre11-Rad50 qui catalyse une étape précoce de la réparation par recombinaison homologue des cassures double brin dans l'ADN. Il a été proposé que le compactage des nucléoïdes fasse partie d'une réponse aux dommages à l'ADN qui accélère la récupération cellulaire en aidant les protéines de réparation de l'ADN à localiser les cibles et en facilitant la recherche de séquences d'ADN intactes lors de la recombinaison homologue.
Voir également
Les références
![]() Cet article a été adapté de la source suivante sous une licence CC BY 4.0 ( 2019 ) ( rapports des évaluateurs ) :
Subhash Verma ; Zhong Qian ; Sankar L Adhya (décembre 2019). « Architecture du nucléoïde Escherichia coli » . PLOS Génétique . 15 (12) : e1008456. doi : 10.1371/JOURNAL.PGEN.1008456 . ISSN 1553-7390 . PMC 6907758 . PMID 31830036 . Wikidata Q84825966 .
Cet article a été adapté de la source suivante sous une licence CC BY 4.0 ( 2019 ) ( rapports des évaluateurs ) :
Subhash Verma ; Zhong Qian ; Sankar L Adhya (décembre 2019). « Architecture du nucléoïde Escherichia coli » . PLOS Génétique . 15 (12) : e1008456. doi : 10.1371/JOURNAL.PGEN.1008456 . ISSN 1553-7390 . PMC 6907758 . PMID 31830036 . Wikidata Q84825966 .

