Splicéosome - Spliceosome
Un spliceosome est un grand complexe de ribonucléoprotéines (RNP) que l'on trouve principalement dans le noyau des cellules eucaryotes . Le spliceosome est assemblé à partir de petits ARN nucléaires ( snRNA ) et de nombreuses protéines (les molécules de petits ARN nucléaires (snRNA) se lient à des protéines spécifiques pour former un petit complexe nucléaire de ribonucléoprotéines (snRNP, prononcé « snurps »), qui à son tour se combine avec d'autres snRNP pour forment un grand complexe ribonucléoprotéique appelé spliceosome) . Le spliceosome supprime les introns d'un pré-ARNm transcrit , un type de transcrit primaire . Ce processus est généralement appelé épissage . Une analogie est un monteur de film, qui découpe sélectivement du matériel non pertinent ou incorrect (équivalent aux introns ) du film initial et envoie la version nettoyée au réalisateur pour le montage final.
Cependant, parfois, l'ARN dans l'intron agit comme un ribozyme, s'épissant sans l'utilisation d'un spliceosome ou d'enzymes protéiques.
Histoire
En 1977, des travaux des laboratoires Sharp et Roberts ont révélé que les gènes d'organismes supérieurs sont "scindés" ou présents en plusieurs segments distincts le long de la molécule d'ADN. Les régions codantes du gène sont séparées par un ADN non codant qui n'est pas impliqué dans l'expression de la protéine. La structure du gène divisé a été trouvée lorsque les ARNm adénoviraux ont été hybridés à des fragments de clivage par endonucléase d'ADN viral simple brin. Il a été observé que les ARNm des hybrides ARNm-ADN contenaient des queues 5' et 3' de régions sans liaison hydrogène. Lorsque des fragments plus grands d'ADN viraux ont été utilisés, des structures fourchues d'ADN en boucle ont été observées lorsqu'elles étaient hybridées aux ARNm viraux. On s'est rendu compte que les régions bouclées, les introns , sont excisées des ARNm précurseurs dans un processus Sharp nommé "épissage". La structure du gène divisé s'est par la suite avérée être commune à la plupart des gènes eucaryotes . Phillip Sharp et Richard J. Roberts ont reçu le prix Nobel de physiologie ou médecine 1993 pour la découverte des introns et le processus d'épissage.
Composition
Chaque spliceosome est composé de cinq petits ARN nucléaires (snRNA) et d'une gamme de facteurs protéiques associés. Lorsque ces petits ARN sont combinés avec les facteurs de protéines, ils font des complexes protéines-ARN appelés snRNPs ( s centre commercial n NUCLÉAIRE r ibo n ucleo p roteins, prononcé « snurps »). Les snRNA qui composent le principal spliceosome sont nommés U1 , U2 , U4 , U5 et U6 , ainsi parce qu'ils sont riches en uridine et participent à plusieurs interactions ARN-ARN et ARN-protéines.
L'assemblage du spliceosome se produit sur chaque pré-ARNm (également connu sous le nom d'ARN nucléaire hétérogène, hn-ARN) à chaque jonction exon:intron. Les introns pré-ARNm contiennent des éléments de séquence spécifiques qui sont reconnus et utilisés pendant l'assemblage des spliceosomes. Ceux-ci comprennent le site d'épissage de l'extrémité 5', la séquence de point de ramification, le tractus polypyrimidine et le site d'épissage de l'extrémité 3'. Le spliceosome catalyse l'élimination des introns et la ligature des exons flanquants.
Les introns ont typiquement une séquence nucléotidique GU au site d'épissage d'extrémité 5' et un AG au site d'épissage d'extrémité 3'. Le site d'épissage 3' peut être défini davantage par une longueur variable de polypyrimidines, appelée polypyrimidine tract (PPT), qui remplit la double fonction de recruter des facteurs sur le site d'épissage 3' et éventuellement de recruter des facteurs sur la séquence de point de branchement (BPS) . Le BPS contient l' adénosine conservée nécessaire à la première étape d'épissage.
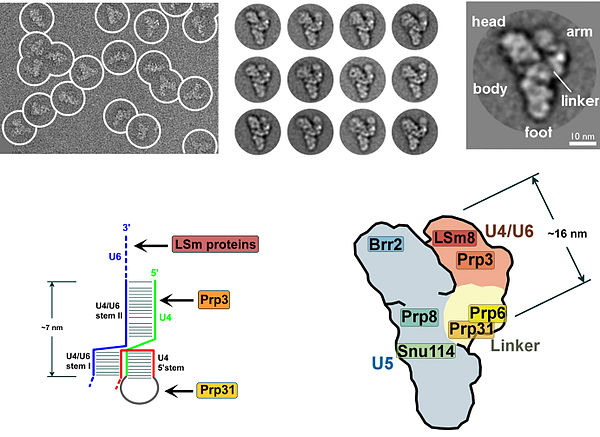
De nombreuses protéines présentent un motif de liaison au zinc, ce qui souligne l'importance du zinc dans le mécanisme d'épissage. La première reconstruction à résolution moléculaire du complexe triple petite ribonucléoprotéine nucléaire (tri-snRNP) U4/U6.U5 a été rapportée en 2016.
La cryo-EM a été largement appliquée par Shi et al pour élucider la structure proche/atomique du spliceosome à la fois chez la levure et chez l'homme. Le cadre moléculaire du spliceosome à une résolution proche de l'atome démontre que le composant Spp42 de U5 snRNP forme un échafaudage central et ancre le centre catalytique dans la levure. La structure atomique du spliceosome humain illustre le composant de l'étape II Slu7 adopte une structure étendue, prête pour la sélection du site d'épissage 3'. Les cinq métaux (assignés comme Mg2+) dans le complexe de levure sont conservés dans le complexe humain.
Épissage alternatif
L'épissage alternatif (la recombinaison de différents exons ) est une source majeure de diversité génétique chez les eucaryotes. Des variantes d'épissage ont été utilisées pour expliquer le nombre relativement faible de gènes codant des protéines dans le génome humain , actuellement estimé à environ 20 000. Un gène particulier de la drosophile , Dscam , a été supposé être épissé alternativement dans 38 000 ARNm différents , en supposant que tous ses exons puissent s'épisser indépendamment les uns des autres.
Assemblée
Le modèle de formation du site actif du spliceosome implique un assemblage ordonné et par étapes de particules snRNP discrètes sur le substrat de pré-ARNm. La première reconnaissance des pré-ARNm implique la liaison du snRNP U1 au site d'épissage de l'extrémité 5' du pré-ARNm et d'autres facteurs non associés au snRNP pour former le complexe d'engagement, ou complexe précoce (E) chez les mammifères. Le complexe d'engagement est un complexe indépendant de l'ATP qui engage le pré-ARNm dans la voie d'épissage. U2 snRNP est recruté dans la région de branche par le biais d'interactions avec le composant du complexe E U2AF (facteur auxiliaire U2 snRNP) et éventuellement U1 snRNP. Dans une réaction dépendante de l'ATP, U2 snRNP devient étroitement associé à la séquence de point de ramification (BPS) pour former le complexe A. Un duplex formé entre U2 snRNP et la région de ramification pré-ARNm fait gonfler la branche adénosine en la spécifiant comme le nucléophile pour le première transestérification.
La présence d'un résidu pseudouridine dans le snRNA U2, presque à l'opposé du site de ramification, entraîne une modification de la conformation du duplex ARN-ARN lors de la liaison U2 snRNP. Plus précisément, la structure altérée du duplex induite par la pseudouridine place le 2' OH de l'adénosine bombée dans une position favorable pour la première étape d'épissage. Le tri-snRNP U4/U5/U6 (voir Figure 1) est recruté dans le spliceosome d'assemblage pour former le complexe B, et après plusieurs réarrangements, le complexe C est activé pour la catalyse. On ne sait pas comment le tri-snRNP est recruté dans le complexe A, mais ce processus peut être médié par des interactions protéine-protéine et/ou des interactions d'appariement de bases entre le snRNA U2 et le snRNA U6.
Le snRNP U5 interagit avec les séquences des sites d'épissage 5' et 3' via la boucle invariante du snRNA U5 et les composants protéiques U5 interagissent avec la région du site d'épissage 3'.
Lors du recrutement du tri-snRNP, plusieurs réarrangements ARN-ARN précèdent la première étape catalytique et d'autres réarrangements se produisent dans le spliceosome catalytiquement actif. Plusieurs des interactions ARN-ARN s'excluent mutuellement ; cependant, on ne sait pas ce qui déclenche ces interactions, ni l'ordre de ces réarrangements. Le premier réarrangement est probablement le déplacement de U1 snRNP du site d'épissage 5' et la formation d'une interaction U6 snRNA. On sait que U1 snRNP n'est que faiblement associé à des spliceosomes complètement formés, et U1 snRNP est un inhibiteur de la formation d'une interaction de site d'épissage U6-5' sur un modèle d'oligonucléotide substrat contenant un exon 5' court et un site d'épissage 5'. La liaison de U2 snRNP à la séquence de point de branchement (BPS) est un exemple d'interaction ARN-ARN déplaçant une interaction protéine-ARN. Lors du recrutement de U2 snRNP, la protéine de liaison de branche SF1 dans le complexe d'engagement est déplacée puisque le site de liaison de U2 snRNA et SF1 sont des événements mutuellement exclusifs.
Au sein du snRNA U2, il existe d'autres réarrangements mutuellement exclusifs qui se produisent entre des conformations concurrentes. Par exemple, sous la forme active, la tige-boucle IIa est privilégiée ; dans la forme inactive, une interaction mutuellement exclusive entre la boucle et une séquence en aval prédomine. On ne sait pas comment U4 est déplacé du snRNA U6, bien que l'ARN ait été impliqué dans l'assemblage des spliceosomes, et puisse fonctionner pour dérouler U4/U6 et favoriser la formation d'une interaction U2/U6 snRNA. Les interactions des tiges-boucles I et II de U4/U6 se dissocient et la région de tige-boucle II libérée de U6 se replie sur elle-même pour former une tige-boucle intramoléculaire et U4 n'est plus nécessaire dans l'assemblage ultérieur des splicéosomes. La région tige-boucle I libérée de U6 paires de bases avec le snRNA U2 formant l'hélice I U2/U6. Cependant, la structure de l'hélice I est mutuellement exclusive avec la moitié 3' d'une région tige-boucle interne 5' du snRNA U2.
Spliceosome mineur
Certains eucaryotes possèdent un second spliceosome, le spliceosome mineur . Un groupe de snRNA moins abondants, U11 , U12 , U4atac et U6atac , ainsi que U5, sont des sous-unités du spliceosome mineur qui épisse une classe rare d'introns pré-ARNm, notés de type U12. Le spliceosome mineur est situé dans le noyau comme son homologue majeur, bien qu'il existe des exceptions dans certaines cellules spécialisées, notamment les plaquettes anucléées et le dendroplasme ( cytoplasme dendrite ) des cellules neuronales.
Les références
Lectures complémentaires
- Boucher SE (2011). "Chapitre 8. Le Spliceosome et ses ions métalliques". Dans Sigel A, Sigel H, Sigel RK (éd.). Rôles structurels et catalytiques des ions métalliques dans l'ARN . Les ions métalliques dans les sciences de la vie. 9 . Éditions RSC. p. 235–51. doi : 10.1039/9781849732512-00235 . ISBN 978-1-84973-094-5. PMID 22010274 .
- Nilsen TW (décembre 2003). "Le spliceosome : la machine macromoléculaire la plus complexe de la cellule ?" . BioEssais . 25 (12) : 1147–9. doi : 10.1002/bies.10394 . PMID 14635248 .
Liens externes
- Structures macromoléculaires 3D des spliceosomes de la banque de données EM (EMDB)
- Spliceosomes à la National Library of Medicine Medical Subject Headings (MeSH) des États-Unis

