Rosetta@home - Rosetta@home
 | |
| Développeur(s) | Laboratoire Baker, Université de Washington ; Rosetta Commons |
|---|---|
| Première version | 6 octobre 2005 |
| Version stable | Rosette : 4,20 / 1er mai 2020
Rosetta Mini : 3,78 / 3 octobre 2017 Rosetta pour Android : 4.20 / 1er mai 2020 |
| Statut de développement | actif |
| Système opérateur | Windows , macOS , Linux , Android |
| Plate-forme | BOINC |
| Licence | Freeware propriétaire pour une utilisation académique et à but non lucratif, licence commerciale disponible |
| Performances moyennes | 487946 Giga FLOPS |
| Utilisateurs actifs | 36 726 |
| Nombre total d'utilisateurs | 1 363 584 |
| Hôtes actifs | 249 673 |
| Nombre total d'hôtes | 529 112 |
| Site Internet | boinc |
Rosetta@home est un projet de calcul distribué pour la prédiction de la structure des protéines sur la plateforme Berkeley Open Infrastructure for Network Computing (BOINC), géré par le laboratoire Baker de l' Université de Washington . Rosetta@home vise à prédire l'amarrage protéine-protéine et à concevoir de nouvelles protéines avec l'aide d'environ cinquante-cinq mille ordinateurs volontaires actifs traitant à plus de 487 946 Giga FLOPS en moyenne au 19 septembre 2020. Foldit , un jeu vidéo Rosetta@home, vise pour atteindre ces objectifs avec une approche de crowdsourcing . Bien qu'une grande partie du projet soit orientée vers la recherche fondamentale pour améliorer la précision et la robustesse des méthodes de protéomique , Rosetta@home fait également de la recherche appliquée sur le paludisme , la maladie d'Alzheimer et d'autres pathologies.
Comme tous les projets BOINC, Rosetta@home utilise les ressources de traitement informatique inactives des ordinateurs des volontaires pour effectuer des calculs sur des unités de travail individuelles . Les résultats complétés sont envoyés à un serveur central du projet où ils sont validés et assimilés dans les bases de données du projet . Le projet est multiplateforme et s'exécute sur une grande variété de configurations matérielles. Les utilisateurs peuvent visualiser la progression de leur prédiction de structure de protéine individuelle sur l'économiseur d'écran Rosetta@home.
En plus de la recherche liée aux maladies, le réseau Rosetta@home sert de cadre de test pour de nouvelles méthodes en bioinformatique structurelle . De telles méthodes sont ensuite utilisées dans d'autres applications basées sur Rosetta, comme RosettaDock ou le projet de pliage du protéome humain et le projet d'immunité au microbiome , après avoir été suffisamment développées et prouvées stables sur l'ensemble vaste et diversifié d'ordinateurs bénévoles de Rosetta@home. Deux tests particulièrement importants pour les nouvelles méthodes développées dans Rosetta@home sont les expériences Critical Assessment of Techniques for Protein Structure Prediction (CASP) et Critical Assessment of Prediction of Interactions (CAPRI), des expériences biennales qui évaluent l'état de l'art dans la structure des protéines. prédiction et prédiction d'amarrage protéine-protéine, respectivement. Rosetta@home se classe systématiquement parmi les principaux prédicteurs d'amarrage et est l'un des meilleurs prédicteurs de structure tertiaire disponibles.
Avec un afflux de nouveaux utilisateurs cherchant à participer à la lutte contre la pandémie de COVID-19 , provoquée par le SARS-CoV-2 , Rosetta@home a augmenté sa puissance de calcul jusqu'à 1,7 PetaFlops au 28 mars 2020. Le 9 septembre. 2020, les chercheurs de Rosetta@home ont publié un article décrivant 10 puissants candidats antiviraux contre le SRAS-CoV-2. Rosetta@home a contribué à cette recherche et ces candidats antiviraux se dirigent vers des essais cliniques de phase 1, qui pourraient commencer début 2022. Selon l'équipe Rosetta@home, les volontaires de Rosetta ont contribué au développement d'un vaccin à nanoparticules. Ce vaccin a été autorisé et est connu sous le nom d' IVX-411 par Icosavax, qui a commencé un essai clinique de phase I/II en juin 2021, et GBP510 qui est développé par SK Bioscience et est déjà approuvé pour un essai clinique de phase III dans le Sud. Corée.
NL-201 , un candidat-médicament anticancéreux qui a d'abord été créé à l'Institute of Protein Design (IPD) et publié dans un article de janvier 2019, a commencé un essai clinique de phase 1 chez l'homme en mai 2021 avec le soutien de Neoleukin Therapeutics, lui-même un spin- hors de l'IPD. Rosetta@home a joué un rôle dans le développement de NL-201 et a contribué à des expériences de « repliement avant » qui ont permis de valider les conceptions de protéines.
Plateforme informatique
L'application Rosetta@home et la plate -forme informatique distribuée BOINC sont disponibles pour les systèmes d'exploitation Windows , Linux et macOS ; BOINC fonctionne également sur plusieurs autres, par exemple FreeBSD. La participation à Rosetta@home nécessite une unité centrale de traitement (CPU) avec une vitesse d'horloge d'au moins 500 MHz , 200 mégaoctets d' espace disque libre , 512 mégaoctets de mémoire physique et une connectivité Internet. Au 20 juillet 2016, la version actuelle de l'application Rosetta Mini est la 3.73. La version actuelle du programme BOINC recommandée est la 7.6.22. Le protocole standard de transfert hypertexte (HTTP) ( port 80) est utilisé pour la communication entre le client BOINC de l'utilisateur et les serveurs Rosetta@home de l'Université de Washington ; HTTPS (port 443) est utilisé lors de l'échange de mot de passe. Le contrôle à distance et local du client BOINC utilise le port 31416 et le port 1043, qui devront peut-être être spécifiquement débloqués s'ils se trouvent derrière un pare - feu . Les unités de travail contenant des données sur des protéines individuelles sont distribuées à partir de serveurs situés dans le laboratoire Baker de l' Université de Washington vers les ordinateurs des volontaires, qui calculent ensuite une prédiction de structure pour la protéine attribuée. Pour éviter les prédictions de structure en double sur une protéine donnée, chaque unité de travail est initialisée avec un numéro de graine aléatoire . Cela donne à chaque prédiction une trajectoire de descente unique le long du paysage énergétique de la protéine . Les prédictions de la structure des protéines de Rosetta@home sont des approximations d'un minimum global dans le paysage énergétique d'une protéine donnée. Ce minimum global représente la conformation la plus énergétiquement favorable de la protéine, c'est-à-dire son état natif .
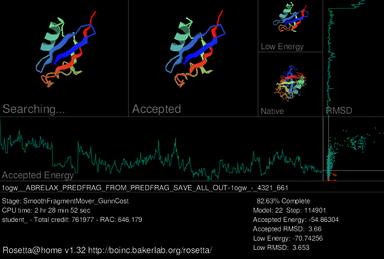
Une caractéristique principale de l' interface utilisateur graphique (GUI) de Rosetta@home est un économiseur d'écran qui montre la progression d' une unité de travail en cours pendant le processus de repliement des protéines simulé . Dans le coin supérieur gauche de l'économiseur d'écran actuel, la protéine cible est montrée adoptant différentes formes (conformations) dans sa recherche de la structure d'énergie la plus basse. Immédiatement à droite se trouve la structure de la plus récemment acceptée. En haut à droite, la conformation d'énergie la plus basse du leurre actuel est affichée ; en dessous se trouve la structure vraie, ou native, de la protéine si elle a déjà été déterminée. Trois graphiques sont inclus dans l'économiseur d'écran. Près du milieu, un graphique pour l' énergie libre thermodynamique du modèle accepté est affiché, qui fluctue au fur et à mesure que le modèle accepté change. Un graphique de l' écart quadratique moyen (RMSD) du modèle accepté, qui mesure à quel point le modèle accepté est structurellement similaire au modèle natif, s'affiche à l'extrême droite. À droite du graphique d'énergie accepté et en dessous du graphique RMSD, les résultats de ces deux fonctions sont utilisés pour produire un graphique énergie vs RMSD au fur et à mesure que le modèle est affiné.
Comme tous les projets BOINC, Rosetta@home s'exécute en arrière-plan de l'ordinateur de l'utilisateur, en utilisant la puissance de l'ordinateur inactif, soit au moment ou avant de se connecter à un compte sur le système d'exploitation hôte . Le programme libère les ressources du processeur car elles sont nécessaires à d'autres applications afin que l'utilisation normale de l'ordinateur ne soit pas affectée. De nombreux paramètres du programme peuvent être spécifiés via les préférences du compte utilisateur, notamment : le pourcentage maximum de ressources CPU que le programme peut utiliser (pour contrôler la consommation d'énergie ou la production de chaleur d'un ordinateur fonctionnant à capacité soutenue), les heures de la journée pendant lesquelles le programme peut s'exécuter , et beaucoup plus.
Rosetta, le logiciel qui s'exécute sur le réseau Rosetta@home, a été réécrit en C++ pour permettre un développement plus facile que celui permis par sa version originale, qui a été écrite en Fortran . Cette nouvelle version est orientée objet et a été publiée le 8 février 2008. Le développement du code Rosetta est réalisé par Rosetta Commons. Le logiciel est sous licence libre pour la communauté universitaire et disponible pour les sociétés pharmaceutiques moyennant des frais.
Importance du projet
Avec la prolifération des projets de séquençage du génome , les scientifiques peuvent déduire la séquence d'acides aminés, ou la structure primaire , de nombreuses protéines qui remplissent des fonctions au sein de la cellule. Pour mieux comprendre la fonction d'une protéine et aider à la conception rationnelle de médicaments , les scientifiques doivent connaître la structure tertiaire tridimensionnelle de la protéine .
Les structures 3D des protéines sont actuellement déterminées expérimentalement par cristallographie aux rayons X ou spectroscopie de résonance magnétique nucléaire (RMN). Le processus est lent (cela peut prendre des semaines, voire des mois pour comprendre comment cristalliser une protéine pour la première fois) et coûteux (environ 100 000 $ US par protéine). Malheureusement, la vitesse à laquelle de nouvelles séquences sont découvertes dépasse de loin la vitesse de détermination de la structure - sur plus de 7 400 000 séquences de protéines disponibles dans la base de données de protéines non redondantes (nr) du National Center for Biotechnology Information (NCBI), moins de 52 000 structures 3D de protéines ont été résolus et déposés dans la Protein Data Bank , le principal référentiel d'informations structurelles sur les protéines. L'un des principaux objectifs de Rosetta@home est de prédire les structures des protéines avec la même précision que les méthodes existantes, mais d'une manière qui nécessite beaucoup moins de temps et d'argent. Rosetta@home développe également des méthodes pour déterminer la structure et l'amarrage des protéines membranaires (par exemple, les récepteurs couplés aux protéines G (RCPG)), qui sont exceptionnellement difficiles à analyser avec des techniques traditionnelles comme la cristallographie aux rayons X et la spectroscopie RMN, mais représentent pourtant la majorité cibles pour les médicaments modernes.
Les progrès dans la prédiction de la structure des protéines sont évalués dans l' expérience semestrielle d' évaluation critique des techniques de prédiction de la structure des protéines (CASP), dans laquelle des chercheurs du monde entier tentent de dériver la structure d'une protéine à partir de la séquence d'acides aminés de la protéine. Les groupes aux scores élevés dans cette expérience parfois compétitive sont considérés comme les porte-drapeaux de facto de l'état de l'art en matière de prédiction de la structure des protéines. Rosetta, le programme sur lequel Rosetta@home est basé, est utilisé depuis CASP5 en 2002. Dans l'expérience CASP6 de 2004, Rosetta est entrée dans l'histoire en étant le premier à produire une prédiction de structure de protéine ab initio proche de la résolution atomique dans son modèle soumis pour la cible CASP T0281. La modélisation ab initio est considérée comme une catégorie particulièrement difficile de prédiction de la structure des protéines, car elle n'utilise pas les informations provenant de l' homologie structurelle et doit s'appuyer sur les informations provenant de l' homologie de séquence et de la modélisation des interactions physiques au sein de la protéine. Rosetta@home est utilisé dans CASP depuis 2006, où il figurait parmi les meilleurs prédicteurs dans toutes les catégories de prédiction de structure dans CASP7. Ces prédictions de haute qualité ont été rendues possibles par la puissance de calcul mise à disposition par les bénévoles de Rosetta@home. L'augmentation de la puissance de calcul permet à Rosetta@home d'échantillonner davantage de régions de l' espace de conformation (les formes possibles qu'une protéine peut prendre), qui, selon le paradoxe de Levinthal , devrait augmenter de façon exponentielle avec la longueur de la protéine.
Rosetta@home est également utilisé dans la prédiction de l' amarrage protéine-protéine , qui détermine la structure de plusieurs protéines complexées , ou structure quaternaire . Ce type d' interaction protéique affecte de nombreuses fonctions cellulaires, y compris la liaison antigène-anticorps et enzyme-inhibiteur ainsi que l'importation et l'exportation cellulaires. La détermination de ces interactions est essentielle pour la conception d'un médicament . Rosetta est utilisé dans l' expérience d' évaluation critique de la prédiction des interactions (CAPRI), qui évalue l'état du champ d'amarrage des protéines de la même manière que les jauges CASP progressent dans la prédiction de la structure des protéines. La puissance de calcul mise à disposition par les volontaires du projet Rosetta@home a été citée comme un facteur majeur des performances de Rosetta dans CAPRI, où ses prévisions d'amarrage ont été parmi les plus précises et les plus complètes.
Au début de 2008, Rosetta a été utilisé pour concevoir informatiquement une protéine avec une fonction jamais observée auparavant dans la nature. Cela a été inspiré en partie par la rétractation d'un article très médiatisé de 2004 qui décrivait à l'origine la conception informatique d'une protéine avec une activité enzymatique améliorée par rapport à sa forme naturelle. Le document de recherche de 2008 du groupe de David Baker décrivant comment la protéine a été fabriquée, qui a cité Rosetta@home pour les ressources informatiques qu'il a mises à disposition, a représenté une preuve de concept importante pour cette méthode de conception de protéines. Ce type de conception de protéines pourrait avoir des applications futures dans la découverte de médicaments, la chimie verte et la bioremédiation .
En plus de la recherche fondamentale sur la prédiction de la structure, de l'amarrage et de la conception des protéines, Rosetta@home est également utilisé dans la recherche immédiate liée aux maladies. De nombreux projets de recherche mineurs sont décrits dans le journal Rosetta@home de David Baker. Depuis février 2014, des informations sur les publications récentes et une brève description des travaux sont mises à jour sur le forum. Le fil du forum n'est plus utilisé depuis 2016, et des nouvelles sur la recherche peuvent être trouvées dans la section des nouvelles générales du projet.
La maladie d'Alzheimer
Un composant de la suite logicielle Rosetta, RosettaDesign, a été utilisé pour prédire avec précision quelles régions des protéines amyloïdogènes étaient les plus susceptibles de produire des fibrilles de type amyloïde . En prenant des hexapeptides (six fragments longs d'acides aminés) d'une protéine d'intérêt et en sélectionnant la plus faible énergie correspondant à une structure similaire à celle d'un hexapeptide connu formant des fibrilles, RosettaDesign a pu identifier des peptides deux fois plus susceptibles de former des fibrilles qu'au hasard. protéines. Rosetta@home a été utilisé dans la même étude pour prédire les structures de la bêta-amyloïde , une protéine formant des fibrilles qui a été postulée pour provoquer la maladie d'Alzheimer . Des résultats préliminaires mais non encore publiés ont été produits sur des protéines conçues par Rosetta qui pourraient empêcher la formation de fibrilles, bien qu'on ne sache pas si elles peuvent prévenir la maladie.
Anthrax
Un autre composant de Rosetta, RosettaDock, a été utilisé en conjonction avec des méthodes expérimentales pour modéliser les interactions entre trois protéines - le facteur létal (LF), le facteur de l'œdème (EF) et l'antigène protecteur (PA) - qui constituent la toxine du charbon . Le modèle informatique a prédit avec précision l'amarrage entre LF et PA, aidant à établir quels domaines des protéines respectives sont impliqués dans le complexe LF-PA. Cette idée a finalement été utilisée dans des recherches qui ont permis d'améliorer les vaccins contre l'anthrax.
Virus de l'herpès simplex 1
RosettaDock a été utilisé pour modéliser l'amarrage entre un anticorps ( immunoglobuline G ) et une protéine de surface exprimée par le virus de l' herpès labial, le virus herpès simplex 1 (HSV-1) qui sert à dégrader l'anticorps antiviral. Le complexe protéique prédit par RosettaDock était étroitement lié aux modèles expérimentaux particulièrement difficiles à obtenir, ce qui a conduit les chercheurs à conclure que la méthode d'amarrage a le potentiel de résoudre certains des problèmes de la cristallographie aux rayons X avec la modélisation des interfaces protéine-protéine.
VIH
Dans le cadre d'une recherche financée par une subvention de 19,4 millions de dollars de la Fondation Bill & Melinda Gates , Rosetta@home a été utilisé pour concevoir plusieurs vaccins possibles contre le virus de l'immunodéficience humaine ( VIH ).
Paludisme
Dans le cadre de recherches menées dans le cadre de l' initiative Grands Défis en santé mondiale , Rosetta a été utilisée pour concevoir par ordinateur de nouvelles protéines endonucléases à tête chercheuse , qui pourraient éradiquer Anopheles gambiae ou rendre le moustique incapable de transmettre le paludisme . Être capable de modéliser et de modifier spécifiquement les interactions protéine-ADN, comme celles des endonucléases à tête chercheuse, confère aux méthodes de conception de protéines informatiques telles que Rosetta un rôle important dans la thérapie génique (qui inclut d'éventuels traitements contre le cancer).
COVID-19 [feminine
La suite de modélisation moléculaire Rosetta a récemment été utilisée pour prédire avec précision la structure à l'échelle atomique de la protéine de pointe du SRAS-CoV-2 des semaines avant qu'elle ne puisse être mesurée en laboratoire. Le 26 juin 2020, le projet a annoncé qu'il avait réussi à créer des protéines antivirales qui neutralisent les virions du SRAS-CoV-2 en laboratoire et que ces médicaments antiviraux expérimentaux sont optimisés pour les essais sur les animaux.
Dans un suivi, un article décrivant 10 inhibiteurs de miniprotéines du SRAS-CoV-2 a été publié dans Science le 9 septembre. Deux de ces inhibiteurs, LCB1 et LCB3, sont plusieurs fois plus puissants que les meilleurs anticorps monoclonaux développés contre le SRAS-CoV. -2, à la fois sur une base molaire et massique. En outre, la recherche suggère que ces inhibiteurs conservent leur activité à des températures élevées, sont 20 fois plus petits qu'un anticorps et ont donc 20 fois plus de sites de neutralisation potentiels, augmentant ainsi l'efficacité potentielle d'un médicament administré localement. La petite taille et la stabilité élevée des inhibiteurs devraient les rendre adéquats pour une formulation de gel qui peut être appliquée par voie nasale ou sous forme de poudre à administrer directement sur le système respiratoire. Les chercheurs travailleront au développement de ces inhibiteurs en thérapeutiques et prophylactiques dans les mois à venir. En juillet 2021, ces candidats antiviraux devaient commencer les essais cliniques au début de 2022 et avaient reçu un financement de la Fondation Bill & Melinda Gates pour des essais précliniques et cliniques précoces. Dans les essais sur les animaux, ces candidats antiviraux étaient efficaces contre des variantes préoccupantes, notamment Alpha, Beta et Gamma.
Rosetta@home a été utilisé pour aider à cribler les plus de 2 millions de protéines de liaison aux pointes du SRAS-CoV-2 qui ont été conçues par ordinateur et ont ainsi contribué à cette recherche.
Selon l'équipe Rosetta@home de l'Institute of Protein Design, les volontaires Rosetta@home ont contribué au développement de candidats médicaments antiviraux et à un vaccin à nanoparticules protéiques. Le vaccin IVX-411 fait déjà l'objet d'un essai clinique de phase 1 mené par Icosavax tandis que le même vaccin, licencié à un autre fabricant et sous le nom GBP510, a été approuvé en Corée du Sud pour un essai de phase III mené par SK Bioscience . Les candidats antiviraux vont également vers des essais cliniques de phase 1.
Cancer
Les chercheurs de Rosetta@home ont conçu un agoniste du récepteur IL-2 appelé Neoleukin-2/15 qui n'interagit pas avec la sous-unité alpha du récepteur. De telles molécules de signal d'immunité sont utiles dans le traitement du cancer. Alors que l'IL-2 naturelle souffre d'une toxicité due à une interaction avec la sous-unité alpha, la protéine conçue est beaucoup plus sûre, du moins dans les modèles animaux. Rosetta@home a contribué à des « expériences de pliage avant » qui ont permis de valider les conceptions.
Dans un article du New Yorker de septembre 2020 , David Baker a déclaré que Neoleukin-2/15 commencerait les essais cliniques sur l'homme « plus tard cette année ». Neoleukin-2/15 est développé par Neoleukin , une entreprise dérivée du laboratoire Baker. En décembre 2020, Neoleukin a annoncé qu'elle soumettrait une demande de nouveau médicament expérimental à la Food and Drug Administration afin de commencer un essai clinique de phase 1 du NL-201, qui est un développement ultérieur de Neoleukin-2/15. Une demande similaire a été soumise en Australie et Neoleukin espère inscrire 120 participants à l'essai clinique de phase 1. L'essai clinique de phase 1 chez l'homme a débuté le 5 mai 2021.
Historique du développement et branches
Initialement introduite par le laboratoire Baker en 1998 en tant qu'approche ab initio de la prédiction de structure, Rosetta s'est depuis diversifiée en plusieurs flux de développement et services distincts. La plate-forme Rosetta tire son nom de la pierre de Rosetta , car elle tente de déchiffrer la "signification" structurelle des séquences d'acides aminés des protéines. Plus de sept ans après la première apparition de Rosetta, le projet Rosetta@home a été publié (c'est-à-dire annoncé comme n'étant plus en version bêta ) le 6 octobre 2005. De nombreux étudiants diplômés et autres chercheurs impliqués dans le développement initial de Rosetta ont depuis déménagé dans d'autres universités. et des instituts de recherche, et par la suite amélioré différentes parties du projet Rosetta.
RosettaDesign
RosettaDesign, une approche informatique de la conception de protéines basée sur Rosetta, a débuté en 2000 avec une étude visant à reconcevoir la voie de repliement de la protéine G . En 2002, RosettaDesign a été utilisé pour concevoir Top7 , une protéine α/β longue de 93 acides aminés qui avait un pli global jamais enregistré auparavant dans la nature. Cette nouvelle conformation a été prédit par Rosetta à moins de 1,2 Å RMSD de la structure déterminée par cristallographie aux rayons X , soit une prédiction de la structure inhabituellement précise. Rosetta et RosettaDesign ont été largement reconnus en étant les premiers à concevoir et à prédire avec précision la structure d'une nouvelle protéine d'une telle longueur, comme en témoigne l'article de 2002 décrivant la double approche qui a suscité deux lettres positives dans la revue Science et cité par plus de 240 autres articles scientifiques. Le produit visible de cette recherche, Top7 , a été présenté comme la « molécule du mois » du RCSB PDB en octobre 2006 ; une superposition des noyaux respectifs (résidus 60-79) de ses structures cristallines prédites et aux rayons X est présentée dans le logo Rosetta@home.
Brian Kuhlman, ancien associé postdoctoral dans le laboratoire de David Baker et maintenant professeur agrégé à l' Université de Caroline du Nord, Chapel Hill , propose RosettaDesign en tant que service en ligne.
RosettaDock
RosettaDock a été ajouté à la suite logicielle Rosetta lors de la première CAPRI expérience en 2002 comme laboratoire de Baker algorithme pour docking protéine-protéine prédiction. Dans cette expérience, RosettaDock a fait une prédiction de haute précision pour l'amarrage entre l' exotoxine pyogène streptococcique A et une chaîne β de récepteur de cellule T , et une prédiction de précision moyenne pour un complexe entre l' α-amylase porcine et un anticorps de camélidé . Alors que la méthode RosettaDock n'a fait que deux prédictions suffisamment précises sur sept possibles, cela suffisait pour la classer septième sur dix-neuf méthodes de prédiction dans la première évaluation CAPRI.
Le développement de RosettaDock a divergé en deux branches pour les rondes CAPRI suivantes alors que Jeffrey Gray, qui a jeté les bases de RosettaDock alors qu'il était à l' Université de Washington , a continué à travailler sur la méthode dans son nouveau poste à l'Université Johns Hopkins . Les membres du laboratoire Baker ont développé RosettaDock en l'absence de Gray. Les deux versions différaient légèrement dans la modélisation de la chaîne latérale, la sélection du leurre et d'autres domaines. Malgré ces différences, les méthodes Baker et Gray ont toutes deux obtenu de bons résultats lors de la deuxième évaluation CAPRI, se classant respectivement cinquième et septième sur 30 groupes de prédicteurs. Le serveur RosettaDock de Jeffrey Gray est disponible en tant que service de prédiction d'amarrage gratuit pour une utilisation non commerciale.
En octobre 2006, RosettaDock a été intégré à Rosetta@home. La méthode utilisait une phase de modèle d'amarrage rapide et brute utilisant uniquement le squelette protéique . Cela a été suivi d'une phase de raffinement lent de l'atome complet au cours de laquelle l'orientation des deux protéines en interaction l'une par rapport à l'autre et les interactions des chaînes latérales à l'interface protéine-protéine ont été simultanément optimisées pour trouver la conformation à plus faible énergie. La puissance de calcul considérablement accrue offerte par le réseau Rosetta@home, combinée à des représentations d' arborescence révisées pour la flexibilité de la dorsale et la modélisation en boucle , a fait de RosettaDock le sixième des 63 groupes de prédiction dans la troisième évaluation CAPRI.
Robetta
Le serveur Robetta (Rosetta Beta) est un service automatisé de prédiction de la structure des protéines proposé par le laboratoire Baker pour la modélisation ab initio et comparative non commerciale . Il a participé en tant que serveur de prédiction automatisé aux expériences CASP semestrielles depuis CASP5 en 2002, se classant parmi les meilleurs dans la catégorie de prédiction de serveur automatisé. Robetta a depuis participé aux CASP6 et 7, où il a fait mieux que la moyenne parmi les groupes de serveurs automatisés et de prédicteurs humains. Il participe également à l' évaluation continue CAMEO3D .
En modélisant la structure des protéines à partir de CASP6, Robetta recherche d'abord des homologues structuraux à l'aide de BLAST , PSI-BLAST et 3D-Jury , puis analyse la séquence cible dans ses domaines individuels , ou des unités de repliement indépendantes des protéines, en faisant correspondre la séquence aux familles structurelles dans la base de données Pfam . Les domaines avec des homologues structuraux suivent ensuite un protocole de "modèle basé sur un modèle" (c'est-à-dire, la modélisation d'homologie ). Ici, le programme d'alignement interne du laboratoire Baker, K*sync, produit un groupe d'homologues de séquence, et chacun d'eux est modélisé par la méthode Rosetta de novo pour produire un leurre (structure possible). La prédiction de structure finale est sélectionnée en prenant le modèle d' énergie le plus bas tel que déterminé par une fonction d'énergie de Rosetta à basse résolution. Pour les domaines qui n'ont pas d'homologues structuraux détectés, un protocole de novo est suivi dans lequel le modèle à plus faible énergie d'un ensemble de leurres générés est sélectionné comme prédiction finale. Ces prédictions de domaine sont ensuite connectées ensemble pour étudier les interactions inter-domaines au niveau tertiaire au sein de la protéine. Enfin, les contributions des chaînes latérales sont modélisées à l'aide d'un protocole de recherche conformationnelle Monte Carlo .
Dans CASP8, Robetta a été augmentée pour utiliser la méthode de raffinement haute résolution de tous les atomes de Rosetta, dont l'absence a été citée comme la principale cause de la moins bonne précision de Robetta que le réseau Rosetta@home dans CASP7. Dans CASP11, un moyen de prédire la carte de contact des protéines par co-évolution de résidus dans des protéines apparentées appelée GREMLIN a été ajouté, permettant plus de succès de novo .
Plie le
Le 9 mai 2008, après que les utilisateurs de Rosetta@home aient suggéré une version interactive du programme informatique distribué , le laboratoire Baker a rendu public Foldit , un jeu de prédiction de structure de protéines en ligne basé sur la plate-forme Rosetta. Au 25 septembre 2008, Foldit comptait plus de 59 000 utilisateurs enregistrés. Le jeu offre aux utilisateurs un ensemble de commandes (par exemple, secouer, agiter, reconstruire) pour manipuler le squelette et les chaînes latérales d' acides aminés de la protéine cible dans des conformations plus énergétiquement favorables. Les utilisateurs peuvent travailler sur des solutions individuellement en tant que solistes ou collectivement en tant qu'évolueurs , accumulant des points dans l'une ou l'autre catégorie à mesure qu'ils améliorent leurs prédictions de structure.
Comparaison avec des projets informatiques distribués similaires
Il existe plusieurs projets de calcul distribué qui ont des domaines d'étude similaires à ceux de Rosetta@home, mais diffèrent dans leur approche de recherche :
Pliage@maison
De tous les grands projets de calcul distribué impliqués dans la recherche sur les protéines, Folding@home est le seul à ne pas utiliser la plate- forme BOINC . Rosetta@home et Folding@home étudient les maladies du repliement des protéines telles que la maladie d'Alzheimer , mais Folding@home le fait beaucoup plus exclusivement. Folding@home utilise presque exclusivement des modèles de dynamique moléculaire de tous les atomes pour comprendre comment et pourquoi les protéines se replient (ou potentiellement mal se replient, puis s'agrègent pour provoquer des maladies). En d'autres termes, la force de Folding@home est de modéliser le processus de repliement des protéines, tandis que la force de Rosetta@home est de calculer la conception des protéines et de prédire la structure et l'amarrage des protéines.
Certains des résultats de Rosetta@home sont utilisés comme base pour certains projets Folding@home. Rosetta fournit la structure la plus probable, mais il n'est pas certain que c'est la forme que prend la molécule ou si elle est viable ou non. Folding@home peut ensuite être utilisé pour vérifier les résultats de Rosetta@home et peut fournir des informations supplémentaires au niveau atomique et des détails sur la façon dont la molécule change de forme.
Les deux projets diffèrent également de manière significative par leur puissance de calcul et la diversité de leurs hôtes. Avec une moyenne d'environ 6 650 téra FLOPS à partir d'une base hôte d' unités centrales de traitement (CPU), d' unités de traitement graphique (GPU) et (anciennement) de PS3 , Folding@home a près de 108 fois plus de puissance de calcul que Rosetta@home.
Réseau communautaire mondial
Les phases I et II du projet de repliement du protéome humain (HPF), un sous-projet de World Community Grid , ont utilisé le programme Rosetta pour effectuer des annotations structurelles et fonctionnelles de divers génomes . Bien qu'il l'utilise maintenant pour créer des bases de données pour les biologistes, Richard Bonneau , scientifique en chef du Human Proteome Folding Project, a été actif dans le développement original de Rosetta au laboratoire de David Baker tout en obtenant son doctorat. Plus d'informations sur la relation entre le HPF1, le HPF2 et Rosetta@home sont disponibles sur le site Web de Richard Bonneau.
Prédicteur@home
Comme Rosetta@home, Predictor@home s'est spécialisé dans la prédiction de la structure des protéines. Alors que Rosetta@home utilise le programme Rosetta pour sa prédiction de structure, Predictor@home a utilisé la méthodologie dTASSER. En 2009, Predictor@home a fermé ses portes.
D'autres projets d'informatique distribuée liés aux protéines sur BOINC incluent QMC@home , Docking@home , POEM@home , SIMAP et TANPAKU . RALPH@home, le projet alpha Rosetta@home qui teste les nouvelles versions d'applications, unités de travail et mises à jour avant de passer à Rosetta@home, fonctionne également sur BOINC.
Contributions bénévoles
Rosetta@home dépend de la puissance de calcul donnée par les membres individuels du projet pour ses recherches. Au 28 mars 2020, environ 53 000 utilisateurs de 150 pays étaient des membres actifs de Rosetta@home, contribuant ensemble au temps d'inactivité du processeur d'environ 54 800 ordinateurs pour une performance moyenne combinée de plus de 1,7 Peta FLOPS .

Les utilisateurs reçoivent des crédits BOINC en tant que mesure de leur contribution. Le crédit accordé pour chaque unité de travail est le nombre de leurres produits pour cette unité de travail multiplié par le crédit moyen réclamé pour les leurres soumis par tous les ordinateurs hôtes pour cette unité de travail. Ce système personnalisé a été conçu pour traiter les différences significatives entre le crédit accordé aux utilisateurs avec le client BOINC standard et un client BOINC optimisé, et les différences de crédit entre les utilisateurs exécutant Rosetta@home sur les systèmes d'exploitation Windows et Linux . Le montant de crédit accordé par seconde de travail CPU est inférieur pour Rosetta@home que la plupart des autres projets BOINC. Rosetta@home est le treizième sur plus de 40 projets BOINC en termes de crédit total.
Les utilisateurs de Rosetta@home qui prédisent les structures protéiques soumises pour l'expérience CASP sont reconnus dans les publications scientifiques concernant leurs résultats. Les utilisateurs qui prédisent la structure énergétique la plus basse pour une unité de travail donnée sont présentés sur la page d' accueil Rosetta@home en tant que Predictor of the Day , ainsi que toute équipe dont ils font partie. Un utilisateur du jour est choisi au hasard chaque jour pour figurer également sur la page d'accueil, parmi les utilisateurs qui ont créé un profil Rosetta@home.
Les références
Liens externes
- Site officiel
- Site Web de Baker Lab Baker Lab
- Le journal Rosetta@home de David Baker
- BOINC Comprend un aperçu de la plate-forme et un guide pour installer BOINC et se connecter à Rosetta@home
- BOINCstats – Rosetta@home Statistiques détaillées des cotisations
- Site Web RALPH@home pour le projet de test alpha Rosetta@home
- Vidéo Rosetta@home sur YouTube Présentation de Rosetta@home donnée par David Baker et les membres du laboratoire
- Rosetta Commons Collaboration académique pour le développement de la plateforme Rosetta
- Page Web du laboratoire Kuhlman , siège de RosettaDesign
Services Rosetta en ligne
- Liste Rosetta Commons des serveurs disponibles
- Serveur de prédiction de structure de protéine Robetta
- ROSIE Docking, design, etc. ensemble de serveurs multifonctions
- Serveur de conception de protéines RosettaDesign
- RosettaBackrub Colonne vertébrale flexible / Serveur de conception de protéines


