Base de données sur les maladies génétiques - Gene Disease Database
| Classification | Bioinformatique |
|---|---|
| Sous-classement | Bases de données |
| Type de bases de données | Biologique |
| Sous-type de bases de données | Gène-Maladie |
En bioinformatique , une base de données sur les maladies génétiques est une collection systématisée de données, généralement structurée pour modéliser des aspects de la réalité, de manière à comprendre les mécanismes sous-jacents de maladies complexes, en comprenant de multiples interactions composites entre les relations phénotype - génotype et les mécanismes gène-maladie. Les bases de données sur les maladies génétiques intègrent des associations gène-maladie humaines provenant de diverses bases de données organisées par des experts et d' associations dérivées de l' exploration de texte, y compris les maladies mendéliennes, complexes et environnementales.
introduction
Des experts de différents domaines de la biologie et de la bioinformatique tentent depuis longtemps de comprendre les mécanismes moléculaires des maladies pour concevoir des stratégies préventives et thérapeutiques. Pour certaines maladies, il est devenu évident que la bonne quantité d'animosité n'est pas suffisante pour obtenir un index des gènes liés à la maladie, mais pour découvrir comment les perturbations des grilles moléculaires dans la cellule donnent lieu à des phénotypes de maladie. De plus, même avec la richesse sans précédent des informations disponibles, l'obtention de tels catalogues est extrêmement difficile.
Génétique D'une manière générale, les maladies génétiques sont causées par des aberrations dans les gènes ou les chromosomes . De nombreuses maladies génétiques se développent avant la naissance. Les troubles génétiques représentent un nombre important des problèmes de santé dans notre société. Les progrès dans la compréhension de ces maladies ont augmenté à la fois la durée de vie et la qualité de vie de nombreuses personnes atteintes de troubles génétiques. Les développements récents de la bioinformatique et de la génétique de laboratoire ont permis de mieux cerner certains syndromes de malformations et de retard mental, afin de comprendre leur mode de transmission. Cette information permet au conseiller génétique de prédire le risque de survenue d'un grand nombre de troubles génétiques. Cependant, la plupart des conseils génétiques ne sont effectués qu'après que la naissance d'au moins une personne affectée a alerté la famille de sa prédilection pour avoir des enfants atteints d'une maladie génétique. L'association d'un seul gène à une maladie est rare et une maladie génétique peut être ou non une maladie transmissible. Certaines maladies génétiques sont héritées des gènes des parents, mais d'autres sont causées par de nouvelles mutations ou modifications de l' ADN . Dans d'autres cas, la même maladie, par exemple certaines formes de carcinome ou de mélanome , peut provenir d'une maladie congénitale chez certaines personnes, de nouveaux changements chez d'autres personnes et de causes non génétiques chez d'autres individus encore.
Il existe plus de six mille troubles monogéniques connus (monogéniques), qui surviennent dans environ 1 naissance sur 200. Comme leur terme le suggère, ces maladies sont causées par une mutation dans un gène. En revanche, les troubles polygéniques sont causés par plusieurs gènes, régulièrement associés à des facteurs environnementaux. Des exemples de phénotypes génétiques comprennent la maladie d'Alzheimer , le cancer du sein, la leucémie, le syndrome de Down, les malformations cardiaques et la surdité ; par conséquent, un catalogage pour trier toutes les maladies liées aux gènes est nécessaire.
Défis avec la création
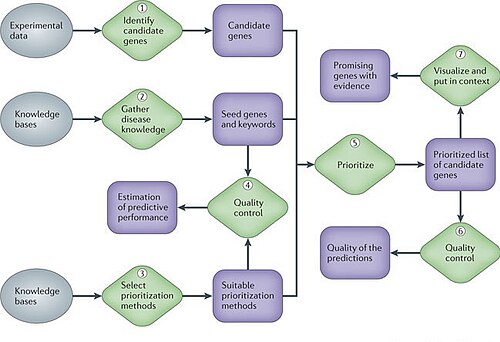
À différentes étapes de tout projet de maladie génétique, les biologistes moléculaires doivent choisir, même après une analyse statistique minutieuse des données, quels gènes ou protéines étudier plus avant expérimentalement et lesquels laisser de côté en raison des ressources limitées. Les méthodes de calcul qui intègrent des ensembles de données complexes et hétérogènes, tels que les données d'expression, les informations de séquence, l'annotation fonctionnelle et la littérature biomédicale, permettent de hiérarchiser les gènes pour une étude future de manière plus éclairée. De telles méthodes peuvent augmenter considérablement le rendement des études en aval et deviennent inestimables pour les chercheurs. Ainsi, l'une des principales préoccupations de la recherche biologique et biomédicale est de reconnaître les mécanismes sous-jacents à ces phénotypes génétiques complexes. De gros efforts ont été consacrés à la recherche des gènes liés aux maladies
Cependant, de plus en plus de preuves indiquent que la plupart des maladies humaines ne peuvent pas être attribuées à un seul gène mais résultent d'interactions complexes entre plusieurs variantes génétiques et facteurs de risque environnementaux. Plusieurs bases de données ont été développées pour stocker des associations entre les gènes et les maladies telles que la base de données de toxicogénomique comparative (CTD), l'héritage mendélien en ligne chez l'homme (OMIM), la base de données d'associations génétiques (GAD) ou la base de données d'associations génétiques de maladies (DisGeNET). Chacune de ces bases de données se concentre sur différents aspects de la relation phénotype-génotype et, en raison de la nature du processus de conservation de la base de données, elles ne sont pas complètes, mais d'une certaine manière, elles sont totalement complémentaires les unes des autres.
Types de bases de données
Il existe essentiellement quatre types de bases de données : les bases de données organisées, les bases de données prédictives, les bases de données bibliographiques et les bases de données intégratives.
Bases de données organisées
Le terme données conservées fait référence à des informations, qui peuvent comprendre les formats de calcul les plus sophistiqués pour les données structurées, les mises à jour scientifiques et les connaissances conservées, qui ont été composées et préparées sous la réglementation d'un ou plusieurs experts considérés comme qualifiés pour s'engager dans une telle activité L'implication est que la base de données résultante est de haute qualité. Le contraste est avec les données qui peuvent avoir été recueillies par le biais d'un processus automatisé ou en utilisant une qualité de données particulièrement faible ou inexperte non prise en charge et peut-être indigne de confiance. Certains des exemples les plus courants incluent : CTD et UNIPROT.
La base de données comparative de toxicogénomique (CTD)
La base de données comparative de toxicogénomique aide à comprendre les effets des composés environnementaux sur la santé humaine en intégrant des données de la littérature scientifique organisée pour décrire les interactions biochimiques avec les gènes et les protéines, et les liens entre les maladies et les produits chimiques, et les maladies et les gènes ou les protéines. CTD contient des données sélectionnées définissant les interactions entre espèces chimiques-gènes/protéines et les associations chimiques et gène-maladie pour éclairer les mécanismes moléculaires sous-jacents à la susceptibilité variable et aux maladies influencées par l'environnement. Ces données fournissent des informations sur les réseaux complexes d'interactions chimiques-gènes et protéines. L'une des principales sources de cette base de données est constituée d'informations organisées par l'OMIM.
CTD est une ressource unique où les spécialistes de la bioinformatique lisent la littérature scientifique et organisent manuellement quatre types de données de base :
- Interactions chimiques-gènes
- Associations de maladies chimiques
- Associations gène-maladie
- Associations chimiques-phénotypes
La ressource protéique universelle (UNIPROT)
L'Universal Protein Resource ( UniProt ) est une ressource inclusive pour les données de séquences protéiques et d'annotation. Il s'agit d'une base de données complète, de première classe et librement accessible de séquences protéiques et d'informations fonctionnelles, qui contient de nombreuses entrées dérivées de projets de séquençage du génome . Il contient une grande quantité d'informations sur la fonction biologique des protéines dérivées de la littérature de l'étude, ce qui peut suggérer un lien direct entre gène-protéine-maladie.
| Teneur | |
|---|---|
| La description | UniProt est la ressource protéique universelle , un référentiel central de données sur les protéines créé en combinant les bases de données Swiss-Prot, TrEMBL et PIR-PSD . |
| Types de données capturés |
Annotation des protéines |
| Organismes | Tous |
| Contact | |
| Centre de recherche | EMBL-EBI , Royaume-Uni ; SIB , Suisse; PIR , États-Unis. |
| Citation principale | Développements en cours et futurs à Universal Protein Resource |
| Accès | |
| Format des données | Fichier plat personnalisé, FASTA , GFF , RDF , XML . |
| Site Internet |
www www |
| URL de téléchargement |
www |
| URL des services Web | Oui - JAVA API voir les informations ici & REST voir les informations ici |
| Outils | |
| la toile | Recherche avancée, BLAST , Clustal O, récupération/téléchargement en masse, mappage d'ID |
| Divers | |
| Licence | Attribution Creative Commons -Pas de modification |
| Gestion des versions | Oui |
Fréquence de diffusion des données |
4 semaines |
| Politique de conservation | Oui – manuel et automatique. Règles d'annotation automatique générées par les conservateurs de bases de données et les algorithmes de calcul. |
Entités pouvant être mises en signet |
Oui - à la fois les entrées de protéines individuelles et les recherches |
Bases de données prédictives
Une base de données prédictive est basée sur l'inférence statistique. Une approche particulière à une telle inférence est connue sous le nom d'inférence prédictive, mais la prédiction peut être entreprise dans l'une des différentes approches de l'inférence statistique. En effet, une description de la biostatistique est qu'elle fournit un moyen de transférer des connaissances sur un échantillon d'une population génétique à l'ensemble de la population ( génomique ) et à d'autres gènes ou génomes connexes, ce qui n'est pas nécessairement la même chose que la prédiction dans le temps. Lorsque les informations sont transférées dans le temps, souvent à des moments précis, le processus est appelé prévision. Trois des principaux exemples de bases de données pouvant être pris en compte dans cette catégorie sont : la base de données du génome de la souris (MGD), la base de données du génome du rat (RGD), l'OMIM et l'outil SIFT de l'Ensembl.
La base de données du génome de la souris (MGD)
La base de données du génome de la souris (MGD) est la ressource de la communauté internationale pour les données génétiques, génomiques et biologiques intégrées sur la souris de laboratoire. MGD fournit une annotation complète des phénotypes et des associations de maladies humaines pour les modèles murins (génotypes) en utilisant les termes de l'ontologie du phénotype des mammifères et les noms des maladies de l'OMIM.
La base de données du génome du rat (RGD)
| Teneur | |
|---|---|
| La description | La base de données du génome du rat |
| Organismes | Rattus norvegicus (rat) |
| Contact | |
| Centre de recherche | Collège médical du Wisconsin |
| Laboratoire | Centre Moléculaire et Génétique Humaine |
| Auteurs | Mary E. Shimoyama, Ph.D. ; Howard J. Jacob, PhD |
| Citation principale | PMID 25355511 |
| Accès | |
| Site Internet | rgd |
| URL de téléchargement | Publication des données RGD |
La base de données du génome du rat (RGD) a commencé comme un effort de collaboration entre les principales institutions de recherche impliquées dans la recherche génétique et génomique du rat. Le rat continue d'être largement utilisé par les chercheurs comme organisme modèle pour étudier la biologie et la physiopathologie de la maladie. Au cours des dernières années, il y a eu une augmentation rapide des données génétiques et génomiques du rat. Cette explosion d'informations a mis en évidence la nécessité d'une base de données centralisée pour collecter, gérer et distribuer efficacement et efficacement une vue centrée sur le rat de ces données aux chercheurs du monde entier. La base de données du génome du rat a été créée pour servir de référentiel de données génétiques et génomiques de rat, ainsi que de cartographie, de souche et d'informations physiologiques. Il facilite également les efforts de recherche des enquêteurs en fournissant des outils pour rechercher, extraire et prédire ces données.
Les données du RGD qui sont utiles aux chercheurs qui étudient les gènes de la maladie comprennent des annotations de la maladie pour les gènes du rat, de la souris et de l'homme. Les annotations sont sélectionnées manuellement à partir de la littérature ou téléchargées via des pipelines automatisés à partir d'autres bases de données liées aux maladies. Les annotations téléchargées sont mappées sur le même vocabulaire de maladie que celui utilisé pour les annotations manuelles afin d'assurer la cohérence dans l'ensemble de données. RGD conserve également des données phénotypiques quantitatives liées à la maladie pour le rat (PhenoMiner).
L'héritage mendélien en ligne chez l'homme ( OMIM )
| Teneur | |
|---|---|
| La description | OMIM est un recueil de gènes humains et de phénotypes génétiques. |
| Organismes | Humain ( H. Sapiens ) |
| Contact | |
| Centre de recherche | NCBI |
| Citation principale | PMID 25398906 |
| Accès | |
| Site Internet | www |
Soutenue par le NCBI, The Online Mendelian Inheritance in Man (OMIM) est une base de données qui répertorie toutes les maladies connues avec une composante génétique, et prédit leur relation avec les gènes pertinents dans le génome humain et fournit des références pour des recherches ultérieures et des outils pour l'analyse génomique. d'un gène catalogué. L'OMIM est un recueil complet et faisant autorité des gènes humains et des phénotypes génétiques, disponible gratuitement et mis à jour quotidiennement. La base de données a été utilisée comme ressource pour prédire les informations pertinentes sur les conditions héritées.
Outil SIFT d'ensemble
 | |
|---|---|
| Teneur | |
| La description | Ensemble |
| Contact | |
| Centre de recherche | |
| Citation principale | Hubbard, et al. (2002) |
| Accès | |
| Site Internet | www |
C'est l'une des plus grandes ressources disponibles pour toutes les études génomiques et génétiques, elle fournit une ressource centralisée aux généticiens, biologistes moléculaires et autres chercheurs étudiant les génomes de notre propre espèce et d'autres vertébrés et organismes modèles de maladies. Ensembl est l'un des nombreux navigateurs génomiques bien connus pour la récupération d'informations sur les maladies génomiques. Ensembl importe des données de variation à partir d'une variété de sources différentes, Ensembl prédit les effets des variantes. Pour chaque variation mappée sur le génome de référence, chaque transcrit Ensembl est identifié qui chevauche la variation. Ensuite, il utilise une approche basée sur des règles pour prédire les effets que chaque allèle de la variation peut avoir sur la transcription. L'ensemble de termes de conséquence, défini par l'ontologie de séquence (SO) peut être actuellement attribué à chaque combinaison d'un allèle et d'un transcrit. Chaque allèle de chaque variation peut avoir un effet différent dans différents transcrits. Une variété d'outils différents sont utilisés pour prédire les mutations humaines dans la base de données Ensembl, l'un des plus largement utilisés est SIFT, qui prédit si une substitution d'acide aminé est susceptible d'affecter la fonction de la protéine sur la base de l'homologie de séquence et de la similitude physico-chimique entre le acides aminés alternatifs. Les données fournies pour chaque substitution d'acide aminé sont un score et une prédiction qualitative (soit « tolérée » ou « délétère »). Le score est la probabilité normalisée que le changement d'acide aminé soit toléré, de sorte que les scores proches de 0 sont plus susceptibles d'être délétères. La prédiction qualitative est dérivée de ce score de telle sorte que les substitutions avec un score < 0,05 sont dites « délétères » et toutes les autres sont dites « tolérées ». SIFT peut être appliqué aux polymorphismes non synonymes naturels et aux mutations faux-sens induites en laboratoire, qui conduiront à établir des relations dans les caractéristiques phénotypiques, la protéomique et la génomique.
Bases de données bibliographiques
Ce type de bases de données résume des livres, des articles, des critiques de livres, des thèses et des annotations sur les bases de données sur les gènes et les maladies. Certains des éléments suivants sont des exemples de ce type : GAD, LGHDN et BeFree Data.
Base de données des associations génétiques (GAD)
La base de données des associations génétiques est une archive d'études d'associations génétiques humaines de maladies complexes. GAD se concentre principalement sur l'archivage d'informations sur les maladies humaines complexes courantes plutôt que sur les troubles mendéliens rares tels qu'ils sont trouvés dans l'OMIM. Il comprend des données résumées extraites d'articles publiés dans des revues à comité de lecture sur les études d'association sur les gènes candidats et le génome ( GWAS ). Le GAD a été gelé au 01/09/2014 mais est toujours disponible en téléchargement.
Réseau gène-maladie humaine dérivé de la littérature (LHGDN)
Le réseau gène-maladie humain dérivé de la littérature (LHGDN) est une base de données dérivée de l'exploration de texte axée sur l'extraction et la classification des associations gène-maladie en fonction de plusieurs conditions biomoléculaires. Il utilise un algorithme basé sur l'apprentissage automatique pour extraire les relations sémantiques gène-maladie à partir d'une source textuelle d'intérêt. Il fait partie des Linked Life Data, du LMU à Munchen, en Allemagne.
Données BeFree
Extrait les associations gène-maladie du résumé MEDLINE à l'aide du système BeFree. BeFree est composé d'un module biomédical de reconnaissance d'entités nommées (BioNER) pour détecter les maladies et les gènes et d'un module d'extraction de relations basé sur des informations morphosyntaxiques.
Bases de données intégratives
Ce type de bases de données inclut les maladies mendéliennes, composées et environnementales dans une archive intégrée d'associations gène-maladie et montre que le concept de modularité s'applique à toutes. Elles fournissent une analyse fonctionnelle des maladies en cas de nouvelles connaissances biologiques importantes, qui pourraient ne pas être découvert en considérant chacune des associations gène-maladie indépendamment. Par conséquent, ils présentent un cadre approprié pour l'étude de la façon dont les facteurs génétiques et environnementaux, tels que les médicaments, contribuent aux maladies. Le meilleur exemple de ce type de base de données est DisGeNET.
La base de données DisGeneNET des associations de maladies génétiques
| Teneur | |
|---|---|
| La description | Intègre les associations gène-maladie humaine |
| Types de données capturés |
Base de données des associations |
| Organismes | Humain ( H. Sapiens ) |
| Contact | |
| Centre de recherche | Programme de Recherche en Informatique Biomédicale (GRIB) IMIM-UPF |
| Laboratoire | Groupe d'informatique biomédicale intégrative |
| Auteurs | Ferran Sanz et Laura I. Furlong (Pinero et al, 2015) |
| Citation principale | PMID 25877637 |
| Accès | |
| Site Internet | www |
| Divers | |
Fréquence de diffusion des données |
annuel |
| Version | 3 |
DisGeNET est une base de données complète d'associations gène-maladie qui intègre des associations de plusieurs sources couvrant différents aspects biomédicaux des maladies. En particulier, il se concentre sur les connaissances actuelles des maladies génétiques humaines, y compris les maladies mendéliennes, complexes et environnementales. Pour évaluer le concept de modularité des maladies humaines, cette base de données effectue une étude systématique des propriétés émergentes des réseaux gène-maladie humains au moyen d'une topologie de réseau et d'une analyse d'annotation fonctionnelle. Les résultats indiquent une origine génétique fortement partagée des maladies humaines et montrent que pour la plupart des maladies, y compris les maladies mendéliennes, complexes et environnementales, des modules fonctionnels existent. De plus, un ensemble de voies biologiques de base s'avère être associé à la plupart des maladies humaines. En obtenant des résultats similaires lors de l'étude de groupes de maladies, les résultats de cette base de données suggèrent que des maladies apparentées pourraient survenir en raison d'un dysfonctionnement de processus biologiques communs dans la cellule. L'analyse du réseau de cette base de données intégrée souligne que l'intégration des données est nécessaire pour obtenir une vue complète du paysage génétique des maladies humaines et que l'origine génétique des maladies complexes est beaucoup plus fréquente que prévu.

La description de chaque type d'association dans cette ontologie est : #Association thérapeutique : Le gène/la protéine a un rôle thérapeutique dans l'amélioration de la maladie. Association #Biomarqueur : Le gène/la protéine joue un rôle dans l'étiologie de la maladie (par exemple, participe au mécanisme moléculaire qui conduit à la maladie) ou est un biomarqueur d'une maladie. #Association de variation génétique : utilisée lorsqu'une variation de séquence (une mutation, un SNP) est associée au phénotype de la maladie, mais qu'il n'y a toujours aucune preuve pour dire que la variation cause la maladie. Dans certains cas, la présence des variants augmente la sensibilité à la maladie. En général, les identifiants NCBI SNP sont fournis. #Association d'expression altérée : les altérations de la fonction de la protéine au moyen d'une expression altérée du gène sont associées au phénotype de la maladie. #Association de modification post-traductionnelle : Des altérations de la fonction de la protéine au moyen de modifications post-traductionnelles (méthylation ou phosphorylation de la protéine) sont associées au phénotype de la maladie.
Quelques cas d'utilisation
Certains des cas les plus intéressants utilisant les bases de données Gene-Disease peuvent être trouvés dans les articles suivants :
- Santiago, José A.; Potashkin, Judith A. (2014). « Une approche en réseau de l'intervention clinique dans les maladies neurodégénératives ». Tendances en médecine moléculaire . 20 (12) : 694–703. doi : 10.1016/j.molmed.2014.10.002 . PMID 25455073 .
- Kaikkonen, Minna U.; Niskanen, Henri; Romanoski, Casey E.; Kansanen, Émilie; Kivelä, Annukka M.; Laitalainen, Jarkko; Heinz, Sven; Benner, Christophe ; Verre, Christopher K.; Ylä-Herttuala, Seppo (2014). "Contrôle des programmes transcriptionnels du VEGF-A par pause et compartimentation génomique" . Recherche sur les acides nucléiques . 42 (20) : 12570–12584. doi : 10.1093/nar/gku1036 . PMC 4227755 . PMID 25352550 .
- Grosdidier, Solène ; Ferrer, Antoni; Faner, Rosa; Piñero, Janet; Roca, Josep; Cosio, Borja; Agustí, Alvar; Gea, Joaquim; Sanz, Ferran ; Furlong, Laura I. (2014). "Analyse médicale en réseau des multimorbidités de la MPOC" . Recherche respiratoire . 15 : 111. doi : 10.1186/s12931-014-0111-4 . PMC 4177421 . PMID 25248857 .
- Cristiano, Francesca; Veltri, Pierangelo (2014). « Un outil basé sur R pour l'analyse des données miARN et la corrélation avec les ontologies cliniques ». Actes de la 5e conférence ACM sur la bioinformatique, la biologie computationnelle et l'informatique de la santé - BCB '14 . p. 768-773. doi : 10.1145/2649387.2660847 . ISBN 9781450328944. S2CID 17123912 .
- Gallagher, Suzanne Renick; Dombrower, Michée ; Goldberg, Debra S. (2014). « En utilisant des coefficients de regroupement d'hypergraphes à 2 nœuds pour analyser les réseaux de gènes de la maladie ». Actes de la 5e conférence ACM sur la bioinformatique, la biologie computationnelle et l'informatique de la santé - BCB '14 . p. 647-648. doi : 10.1145/2649387.2660817 . ISBN 9781450328944. S2CID 30593231 .
- Mannil, Deepthi ; Vogt, Ingo; Prinz, Jeanette ; Campillos, Monique (2015). « BD d'hétérogénéité du système d'organes : une base de données pour la visualisation des phénotypes au niveau du système d'organes » . Recherche sur les acides nucléiques . 43 (problème de base de données) : D900–D906. doi : 10.1093/nar/gku948 . PMC 4384019 . PMID 25313158 .
- Vogt, Ingo; Prinz, Jeanette ; Campillos, Monica (2014). "Les médicaments et les maladies moléculairement et cliniquement liés sont enrichis en paires médicament-maladie phénotypiquement similaires" . Médecine du génome . 6 (7) : 52. doi : 10.1186/s13073-014-0052-z . PMC 4165361 . PMID 25276232 .
- Santiago, José A.; Potashkin, Judith A. (2014). « Approches systémiques pour décoder les liens moléculaires dans la maladie de Parkinson et le diabète ». Neurobiologie de la maladie . 72 : 84-91. doi : 10.1016/j.nbd.2014.03.019 . PMID 24718034 . S2CID 41944859 .
- Lee, In-Hee ; Lee, Kyungjoon ; Hsing, Michel ; Choe, Yongjoon ; Parc, Jin-Ho ; Kim, Shu Hee; Bohn, Justin M.; Neu, Matthieu B.; Hwang, Kyu-Baek ; Vert, Robert C.; Kohane, Isaac S.; Kong, Sek Won (2014). "Prioriser les variantes, les gènes et les voies liés à la maladie avec un pipeline d'analyse interactive du génome entier" . Mutation humaine . 35 (5) : 537-547. doi : 10.1002/humu.22520 . PMC 4130156 . PMID 24478219 .
- Liu, Ming-Xi ; Chen, Xing ; Chen, Geng ; Cui, Qing-Hua ; Yan, Gui-Ying (2014). "Un cadre de calcul pour déduire les ARN longs non codants associés aux maladies humaines" . PLOS UN . 9 (1) : e84408. Bibcode : 2014PLoSO ... 984408L . doi : 10.1371/journal.pone.0084408 . PMC 3879311 . PMID 24392133 .
- Zhao, Yilei ; Wang, Chen ; Wu, Jianwei ; Wang, Yan ; Zhu, Wenliang ; Zhang, Yong ; Du, Zhimin (2013). "La choline protège contre l'hypertrophie cardiaque induite par une post-charge accrue" . Journal international des sciences biologiques . 9 (3) : 295-302. doi : 10.7150/ijbs.5976 . PMC 3596715 . PMID 23493786 .
- Koczor, Christophe A.; Lee, Eva K.; Torres, Rebecca A.; Boyd, Amy; Vega, J. David; Uppal, Karan ; Yuan, Fan ; Champs, Earl J.; Samarel, Allen M. ; Lewis, Guillaume (2013). « Détection de promoteurs de gènes différentiellement méthylés dans le myocarde du ventricule gauche humain défaillant et non défaillant à l'aide d'une analyse informatique » . Génomique physiologique . 45 (14) : 597-605. doi : 10.1152/physiolgenomics.00013.2013 . PMC 3727018 . PMID 23695888 .
- Gu, Ying ; Liu, Guang Hui ; Plongthongkum, Nongluk ; Benner, Christophe ; Yi, Fei; Qu, Jing ; Suzuki, Keiichiro ; Yang, Jiping ; Zhang, Weiqi ; Li, Mo ; Montserrat, Nuria ; Crespo, Isaac ; Del Sol, Antonio ; Esteban, Concepcion Rodriguez; Zhang, Kun ; Izpisua Belmonte, Juan Carlos (2014). "Méthylation globale de l'ADN et analyses transcriptionnelles des cardiomyocytes dérivés de l'ESC humain" . Protéine & Cellule . 5 (1) : 59-68. doi : 10.1007/s13238-013-0016-x . PMC 3938846 . PMID 24474197 .
- Galhardo, Mafalda; Sinkkonen, Lasse; Berninger, Philippe ; Lin, Jacques ; Sauter, Thomas ; Heinäniemi, Merja (2014). "L'analyse intégrée de la régulation du métabolisme au niveau de la transcription révèle des nœuds pertinents pour la maladie du réseau métabolique humain" . Recherche sur les acides nucléiques . 42 (3) : 1474-1496. doi : 10.1093/nar/gkt989 . PMC 3919568 . PMID 24198249 .
- Tieri, Paulo ; Termanini, Alberto; Bellavista, Elena ; Salvioli, Stefano; Capri, Myriam ; Franceschi, Claudio (2012). " Cartographier la carte d'interaction de la voie NF-κB " . PLOS UN . 7 (3) : e32678. Bibcode : 2012PLoSO ... 732678T . doi : 10.1371/journal.pone.0032678 . PMC 3293857 . PMID 22403694 .
Remarques sur l'avenir dans les bases de données sur les maladies des gènes
L'achèvement du génome humain a changé la façon dont la recherche des gènes de la maladie est effectuée. Dans le passé, l'approche consistait à se concentrer sur un ou quelques gènes à la fois. Aujourd'hui, des projets comme le DisGeNET illustrent les efforts déployés pour analyser systématiquement toutes les altérations génétiques impliquées dans une ou plusieurs maladies. L'étape suivante consiste à produire une image complète des aspects mécanistiques des maladies et de la conception de médicaments contre celles-ci. Pour cela, une combinaison de deux approches sera nécessaire : une recherche systématique et une étude approfondie de chaque gène. L'avenir du domaine sera défini par de nouvelles techniques permettant d'intégrer de grands ensembles de données provenant de différentes sources et d'incorporer des informations fonctionnelles dans l'analyse de données à grande échelle générées par des études bioinformatiques.
La bioinformatique est à la fois un terme désignant l'ensemble des études sur les maladies génétiques biologiques qui utilisent la programmation informatique dans le cadre de leur méthodologie, ainsi qu'une référence à des pipelines d'analyse spécifiques qui sont utilisés à plusieurs reprises, en particulier dans les domaines de la génétique et de la génomique. Les utilisations courantes de la bioinformatique comprennent l'identification de gènes et de nucléotides candidats, les SNP . Souvent, une telle identification est faite dans le but de mieux comprendre la base génétique de la maladie, les adaptations uniques, les propriétés souhaitables ou les différences entre les populations. De manière moins formelle, la bioinformatique essaie également de comprendre les principes d'organisation au sein des séquences d'acides nucléiques et de protéines.
La réponse de la bioinformatique aux nouvelles techniques expérimentales apporte une nouvelle perspective dans l'analyse des données expérimentales, comme le démontrent les progrès de l'analyse des informations provenant des bases de données sur les maladies génétiques et d'autres technologies. On s'attend à ce que cette tendance se poursuive avec de nouvelles approches pour répondre aux nouvelles techniques, telles que les technologies de séquençage de nouvelle génération. Par exemple, la disponibilité d'un grand nombre de génomes humains individuels favorisera le développement d'analyses informatiques de variantes rares, y compris l'exploration statistique de leurs relations avec les modes de vie, les interactions médicamenteuses et d'autres facteurs. La recherche biomédicale sera également guidée par notre capacité à exploiter efficacement le vaste corpus de données biomédicales existantes et générées en continu. Les techniques d'exploration de texte, en particulier, lorsqu'elles sont combinées avec d'autres données moléculaires, peuvent fournir des informations sur les mutations et les interactions génétiques et deviendront cruciales pour garder une longueur d'avance sur la croissance exponentielle des données générées dans la recherche biomédicale. Un autre domaine qui bénéficie des progrès de l'exploitation minière et de l'intégration de l'analyse moléculaire, clinique et médicamenteuse est la pharmacogénomique. Les études in silico des relations entre les variations humaines et leurs effets sur les maladies seront essentielles au développement de la médecine personnalisée. En résumé, les bases de données génétiques sur les maladies ont déjà transformé la recherche de gènes de maladies et pourraient devenir un élément crucial d'autres domaines de la recherche médicale.
Voir également
- Informatique de la biodiversité
- Entreprises de bioinformatique
- Biomédecine
- Biologie computationnelle
- Biomodélisation computationnelle
- Génomique computationnelle
- Identification des gènes de la maladie
- Institut Européen de Bioinformatique
- Génomique fonctionnelle
- Informatique de la santé
- Projet du génome humain
- Bioinformatique intégrative
- Société internationale de biologie computationnelle
- Bibliothèque de saut
- Liste des revues de bioinformatique
- Liste des bases de données biologiques
- Liste des logiciels de bioinformatique open source
- Pathologie
- Phylogénétique
- Bioinformatique structurelle


